BLAST
| BLAST
| |
|---|---|
 | |
| Nasljednik | FASTA-e, iz 1985 |
| Datum osnivanja | 1990. |
| Vrsta | Baza genomskih podataka |
| Status | Aktivna |
| Glavno sjedište | NCBI, SAD |
| Jezik | Engleski |
| Br. volontera | Neograničen |
| Veb-sajt | blast |
U bioinformatici, BLAST (eng. Basic Local Alignment Search Tool) je algoritam za poređenje primarnih bioloških sekvenci, kao što su aminokiseline raznih proteina ili nukleotid a u molekulama DNK.[1] Pretraživač BLAST pomogućava spoređenje mnoštva sekvenci sa bibliotekama ili bazama podataka o sekvencama i identificiraju sekvencu koja odgovara traženoj, sa određenom greškom.
BLAST-u je The New York Times nazivao Googleom bioloških istraživanja i jedan je od najčešće korištenih bioinformatičkih programa za pretraživanje sekvenci.[2]
Danas su dostupni raazličiti tipovi BLAST-a, u zavisnosti od tipa pretraživane sekvence. Naprimjer, nakon otkrića prethodno nepoznatog gena kod miševa, obično se uključuje BLAST- pretraga ljudskih genoma u provjeri da li dati ljudi sadrže slične gene, BLAST prepoznaje sekvence u genomu koje liče mišjem, zasnovanom na sličnosti sekvenci. BLAST-ov algoritam i program dizajnirali su i Stephen Altschul, Warren Gish, Webb Miller, Eugene Myers, i David J. Lipman u Nacionalnom institutu za zdravlje Sjedinjenih Država i objavili u Journal of Molecular Biology, 1990.[3]
Okviri[uredi | uredi izvor]
BLAST je jedan od najčešće koršćenih programa u bioinformatici za pretragu sekvenci.[4] Predstavlja temelj za rješavanje problem u bioinformatici. Korištemi Heuristički algoritam je mnogo brži nego drugi pristupi, kao što je računanje optimalnog poravnanja. Naglasak na brzini je ključan aspekat algoritma, posebno na velikim sada dostupnim bazama genoma, iako noviji algoritmi mogu biti još brži. Za slične potrebe, prije BLAST-a, David J. Lipman i William R. Pearson kreirali su FASTA, 1985.[5] Prije brzih algoritama, kao što su BLAST i FASTA, pretraživbanja proteina i nukleinskih sekvenci bila je vremenski veoma zahtijevna jer je korišten potpuni postupak poravananja (npr., Smit-Vatermanov algoritam).
Iako je BLAST brži od svih Smith-Watermanovih implementacija za većinu slučajeva, ipak ne može "garantirati optimalno poravnavanje upita sa bazom sekvenci" kao Smith-Watermanov. Optimalnost S–W algoritma "obezjbeđuje najveću tačnost i najpreciznije rezultate", po cijenu vremena i resursa računara.
BLAST je vremenski efikasniji od FASTAjer budući da pretražuje samo značajnije uzorake u sekvenci, ali sa potrebnom osetljivošću.
Primjeri upotrebe BLAST-a, između ostalih, je traženje odgovora na upite:
- Koje vrste bakterija imaju protein koji je u vezi sa određenim proteinom sa poznatom aminokiselinom?
- Koji još geni predstavljaju proteine koji imaju strukturu sličnu onima koji su već otkriveni?
BLAST je često korišćen kao komponenta drugih algoritama koji zahtijevaju približno poklapanje sekvenci.
Ulaz[uredi | uredi izvor]
Ulaz predstavljaju sekvence (u FASTA formatu ili Genbank formatu) i težinsku matricu.
Izlaz[uredi | uredi izvor]
Izlaz BLAST algoritma može biti predstavljen na različite načine. Korišteni formati mogu biti HTML, tekst i XML. Za web stranicu NCBI, podrazumijevani format izlaza je HTML. Kada se BLAST algoritam realizira na NCBI sajtu, rezultati se dobiju u grafičkom obliku i prikazuju pogotke, a tabele p identifikatore sekvenci za pogotke, zajedno sa pratećim podacima, kao i poravnanje sekvenci od interesa. Uključuje i pogotke dobijene korišćenjem odgovarajućeg vrijednosnog sistema BLAST. su Tabele su i najjednostavnije i najinformativnije za čitanje.
Ako se traži sekvenca koje nema u javno dostupnim bazama putem izvora, kao što je NCBI sajt, BLAST-ov algoritam je moguće besplatno preuzeti sa interneta. Može se preuzeti sa NCBI sajta[6]. Dostupni su i komercijalni programi, a baze se mogu naći na NCBI sajtu, kao i na indeksu BLAST baza podataka[7].
Proces[uredi | uredi izvor]
Korištenjem heurističkih metoda, BLAST nalazi slične sekvence, lociranjem kratkih poklapanja između poređenih sekvenci. Ovaj proces pronalaženja naziva se sijanje (eng.seeding). Nakon prvog poklapanja, BLAST počinje praviti lokalna poravnavanja. Dok pokušava naći sličnost u sekvenci, veoma je važan skup čestih slova, poznat kao riječ. Naprimjer, ako sekvenca sadrži niz slova: GLKFA. Ako se BLAST pokrene pod normalnim uvjetima, riječ bi bila duga tri slova. U ovom slučaju, korištenjem datog niza slova, dobijene riječi bi bile GLK, LKF, KFA. BLAST-ov heuristički algoritam locira sve česte pojave troslovnih riječi između zadate i pronađene sekvence. Ovaj rezultat se zatim koristi za kreiranje poravnavanja. Nakon što je napravio moguće riječi za posmatranu sekvencu, također su obrađene i ostale. Te riječi, u poređenju sa matricom vrijednosti, moraju imati zadovoljen prag T. Često korištena matrica vreijdnosti za BLAST pretraživanja je BLOSUM62, iako optimalna matrica vrijednosti ovisi od sličnosti sekvenci. Kada su riječi, kao i okolne , procesuirane, porede se sa sekvencama iz baze u cilju pronalaska poklapanja. O tome da li konkretna riječ ulazi u poravnanje određuje Prag T. Kada se obavi sijanje , poravnanje dužine 3, prošireno je u oba smjera pomoću BLAST algoritma. Svako proširenje utiče na rezultat poravnanja, bilo povećanjem bilo smanjenjem. Ako je rezultat veći od unaprijed određenog T, poravnanje će biti uključeno u rezultat BLAST-a. Međutim, ako je rezultat manji od predodređenog T, poravnanje će prestati da se širi, sprečavajući da se segmenti sa lošim poravnanjem uključe u rezultat BLAST-a. Treba uočiti da dse povećanjem T ograničava prostor koji se može pretraživati i smanjujej broj susednih riječi, dok se istovremeno ubrzava BLAST.[8]
Algoritam[uredi | uredi izvor]
Za pokretanje programa, BLAST zahtijeva unos sekvence za pretragu i sekvence sa kojom će upoređivati (također znava i “ciljana sekvenca”) ili baze koja sadrži više sekvenci. BLAST će naći više podsekvenci u bazi koje su slične podsekvenci upita. Obično, upitna sekvenca je dosta manja od baze, naprimjer, upit može sadržati hiljadu nukleotida, dok baza ih sadrži nekoliko milijardi.
Glavna ideja BLAST-a je da često postoji visoko rangirani segmentni parovi (High-scoring Segment Pairs – HSP) sadržani u statistički bitnom poravnanju. BLAST traži visoko rangirana poravnanja između upitne i posmatrane sekvence iz baze, koristeći heuristički pristup, koji aproksimira Smith-Waterman algoritam. Međutim, iscrpni Smith-Watermanov pristup suviše je spor za pretraživanje velikih baza genoma, kao što je GenBank. Zato se BLAST algoritam koristi heuristički i manje je precizan od Smith-Watermanovog algoritma, ali preko 50 puta brži. Brzina i relativno dobra preciznost BLAST-a su među ključnim tehničkim inovacijama BLAST programa. Pregled BLAST algoritma (protein-protein pretraga):[9] and CTGA2016
- Brisanje nekompleksnih regija ili ponavljajućih sekvenci iz upita.
- Nekompleksirana regija predstavlja područje sekvenci koje je sastavljeno od nekoliko tipova elemenata. Ove regije mogu onemogućiti program da nađe bitnu sekvencu u bazi, pa bi ih trebalo isfiltrirati. Te regije se obilježe sa X (proteinske sekvence) ili N (nukleinske kiseline) i biće ignorirane u programu BLAST-a. Za filtriranje nekompleksnih regija proteinskih sekvenci koristi se program SEG[10], a za DNK sekvence program DUST[11].
- Pravljenje k-slovne liste reči upitne sekvence.
- Ako se uzme k=3 kao primjer, izlistavaju se riječi dužine 3 iz upitne proteinske sekvence (k je obično 11 za sekvencuDNK ) "sekvencino", dok ne budu uključena sva slova upitne sekvence. Metod je prikazan na slici ispod.

- Lista mogućih riječi koje se poklapaju.
- Ovaj korak je jedna od glavnih razlika između BLAST-a i FASTA-a. FASTA koristi sve česte riječi nađene u koraku 2; ali BLAST jedino uzima visokovrednovane riječi. Vrijednosti su kreirane upoređivanjem riječi dobijenih u koraku 2 sa svim preostalim. Korištenjem matrice vrijednosti i poređenjem svih parova, postoji 203 mogućih komcinacija za troslovne riječi. Naprimjer, vrijednost koja se dobije poređenjem PQG sa PEG i PQA je 15 i 12, redom. Za riječi u DNK, pogodak se računa kao +5, a promašaj kao –4, ili kao +2 i –3. Nakon toga, vrijednost praga susjednih riječi T koristi se da smanji broj mogućih riječi koje se poklapaju. One riječi čije su vrijednosti veće od praga T ostat će na listi tih riječi, dok će one čija je vrijednost manja od ovog praga biti odbačene. Naprimjer, kada je T = 13, PEG se čuva, ali PQA je odbačen.
- Organizacija visokorangiranih riječi u efikasno stablo pretrage.
- Ovo omogućava programu da brzo poredi visoko rangirane reči sa onima iz baze.
- Ponavljanje koraka 3 – 4 za svaku k-slovnu riječ iz upitne sekvence.
- Pretraga baze za pronalaženja egzaktnog poklapanja sa preostalim visokorangiranim riječima.
- BLAST program pretražuje bazu po preostalim visokorangiranim riječima, kao što je PEG, po svakoj poziciji. Ako se pronađe egzaktno poklapanje, koristi se za sijanje mogućih poravnanja bez odstupanja između upitne sekvence i sekvence iz baze.
- Proširivanje egzaktnog poklapanja u visoko rangirani segmentni par (HSP).
- Originalna verzija BLAST-a pravi dugo poravnanje između upita i sekvenci iz baze u oba smjera od pozicije gde je pronađeno egzaktno poklapanje. Proširenje ne prestaje sve dok nagomilana vrijednost HSP-a ne počne opadati.
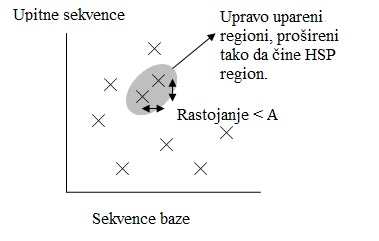
- Radi uštede vremena, razvijena je novija verzija BLAST-a, zvana BLAST2. BLAST2 prisvaja nižerangirane susjedne riječi da bi očuvao isti nivo osetljivosti za otkrivanje sličnih sekvenci. Zato lista mogućih poklapanja iz koraka 3 postaje duža. Nadalje, regije kod kojih je došlo do egzaktnog poklapanja, na međusobnom rastojanju A na istoj dijagonali na gornjoj slici, biće spojeni u jednu dugu novu regiju. Konačno, nove regije su proširene na isti način kao u originalnoj verziji BLAST-a.
- Originalna verzija BLAST-a pravi dugo poravnanje između upita i sekvenci iz baze u oba smjera od pozicije gde je pronađeno egzaktno poklapanje. Proširenje ne prestaje sve dok nagomilana vrijednost HSP-a ne počne opadati.
- Lista svih HSP u bazi čija je vrijednost dovoljno velika da se uzmu u obzir.
- Zatim se listaju vrijednosti HSP-a koje su veće od empirijski utvrđenih granica vrednosti S. Proučavanjem distribucije vrijednosti poravnanja, dobijenih poređenjem slučajnih sekvenci, granična vrijednost S može biti utvrđena tako da njena vrijednost bude dovoljno velika da garantira značajan ostatak HSP-a.
- Procjena značajnosti vrijednosti HSP-a
- BLAST dalje pristupa statističkom vrednovanju svake HSP vrijednosti, korišćenjem Gumbelove distribucije ekstremnih vrijednosti (EVD)[12]. U poređenju sa Gumbel EVD, verovatnoća p za dostizanje S, koja je veća ili jednaka od x, data je sledećom jednačinom:
- gdje je
- Statistički parametri i procjenjuju se na osnovu vriednosti poravnanja bez rupa. Tako da i zavise od matrice zamjene i kompozicije sekvence. i su efektivne dužine unosa i sekvence iz baze, redom. Dužina sekvence je skraćena na efektivnu dužinu, da bi kompenzirala rubni efekt. Mogu se izračunati kao:
- gdje je očekivana srednja vrijednost po poravnatom paru u poravnanju dvije slučajne sekvence. Altschul i Gish dali su uobičajene vrednosti, , , i , za lokalno poravnanje bez rupa, korišćenjem BLOSUM62[13] kao zamjenske matrice. Korištenje tipskih vrijednosti za procjenu značajnosti se zove metod table sa pogledom nagore, koji nije precizan. Očekivana vrijednost E je broj koliko puta nepovezana sekvenca iz baze , po zakonu vjerovatnoće, dobiti vrijednost S veću od x. Očekivano E, koje je dobijeno u pretrazi baze D sekvenci, dato je obrascem:
- Štaviše, kada je , E može biti aproksimirano pomoću Poissonove distribucije, kao
- Očekivana vrijednost E dodeljuje značaj vrijednostima HSP-a za lokalno poravnanje bez rupa, i to se predstavlja kao rezultat BLAST-a. Izračunavanja prikazana ovdje se modificiraju, ako se pojedinačni HSP-ovi kombiniraju kao da predstavljaju rupičasto poravnanje, zbog varijacije statističkih parametara.
- Spajanje dvije ili više HSP-regija u duže poravnanje.
- Ponekad se nađu dvije ili više HSP-regija u jednoj sekvenci iz baze, koji se mogu spojiti u duže poravnanje. Ovo omogućava dodatnu evidenciju o relacijama između upita i sekvence iz baze. U tom pracvcu, postoje dva metoda: Poissonov metod i rezultat sume kvadrata, za poređenje značajnosti novodobijenih HSP-regija. Ako se pretpostavi postojanje dvije kombinirane HSP-regije sa vrijednostima (65, 40) i (52, 45), tada Poissonov metod pridaje veći značaj skupu sa maksimalnom nižom vrednošću (45>40). Međutim, metod sume kvadrata daje veći značaj prvom skupu, zato što je 65+40 (105) veće od 52+45(97). Originalni BLAST koristi Poissonov metod; BLAST2 i WU-BLAST primjenjuju metod sume kvadrata.
- Prikaz ispražnjenog Smith-Watermanovog lokalnog poravnanja u upitu i uparenih sekvenci iz baze.
- Originalni BLAST generira samo cijela poravnanja, uključujući inicijalno uspostavljene HSP, čak i ako ima više od jednog HSP-a u sekvenci iz baze.
- BLAST2 koristi jednostruko rupičasto poravnanje koje uključuje sve inicijalno pronađene HSP regije.
- Pokaz svakog pogotka čija je očekivana vrijednost manja od parametra E.
Paralelni BLAST[uredi | uredi izvor]
Verzija paralelnog BLAST-a, koja koristi razdvojene baze implementirana je korištenjem MPI i Pthreads, i prilagođena je različitim platformama, uključijući i Windows, Linux, Solaris, Mac OS X i AIX. Popularni pristup paraleliziranja BLAST-a uključuje distribuirane upite, segmentaciju keš tabela, paralelno računanje i segmentaciju baza. Baze su podeljene na jednake dijelove i čuvaju se na lokalnim čvorovima. Svaki upit pokrenut na svim čvorovima paralelno je i izlazni za fajlove koji su spojeni u finalni izlaz.[14]
Program[uredi | uredi izvor]
Program BLAST-a može biti, ili preuzet, ili pokrenut iz komandne linije, ili se može koristiti besplatno onlajn. BLAST-ov web server, kojeg održava NCBI, dozvoljava svakome sa web pretraživačem da obavlja slične pretrage na konstantno ažuriranoj bazi proteina i DNK, koja uključuje većinu organizama. Ovaj program je otvorenog koda, što daje svima mogućnost da ga i koriste i mijenjaju. To je dovelo do nastanka više varijanti programa BLAST.
Danas su dostupne različite korisne varijacije BLAST-a, koje mogu biti upotrebljene u zavisnosti od onoga šta se želi raditi i čime se radi. Ove varijacije programa su različite po upitnim sekvencama baze koja se pretražuje i šta se upoređuje. Ovi programi i njihovi opisi su izlistani ispod: BLAST je zapravo porodica programa (sve su uključene u Blastall ). Ovo uključuje:[15]
- Nukleotid-nukleotid BLAST (blastn) je program, kojem se zadaje upit o DNK, a vraća najsličniju DNK sekvencu iz baze koju je korisnik odabrao.
- Protein-protein BLAST (blastp)
- Ovaj program, kojem se zadaje proteinski upit, vraća najsličniju proteinsku odabranu sekvencu iz baze .
- Pozicijski-specifični interktivni BLAST (PSI-BLAST) (blastpgp) koristi se za pronalaženje daljih srodnika proteina. Prvo se kreira lista svih blisko povezanih proteina. Ovi proteini se kombiniraju u opću “profilnu" sekvencu, koja sumira značajne obilježja u tim sekvencama. Upit na bazi proteina koristi taj profil i pronalazi veću grupu proteina. Ova veća grupa koristi se za pravljenje novog profila i proces se ponavlja. Blast [16] is an accelerated version of NCBI BLASTP for CUDA which is 3x~4x faster than NCBI Blast.
- Uključivanjem povezanih proteina u pretragu, PSI-BLAST je osetljiviji u izboru srodnika iz filogenetskog stabla, od običnog protein-protein BLAST-a.
- Nukleotid]ni 6-okvir translacije proteina (blastx) poredi šest okvira koncepta translacije nukleotida upitne sekvence sa proteinom iz baze.
- Nukleotidna 6-okvirna translacija nukleotida 6-okvirnom translacijom (tblastx) je najsporiji program BLAST-ove porodice. Translira upitnu sekvencu nukleotida u svih šest mogućih okviraa i poredi ih sa šest okvira, translacijom nukleotida upitne sekvence sa proteinom iz baze. Svrha ovoga je da nađe veoma daleke veze između nukleotidnih sekvenci.
- Protein-nukleotidna 6-okvirna translacija (tblastn) poredi upit sa svih šest okvira sa sekvencom nukleotida iz baze.
- Veliki broj upitnih sekvenci (megablast)
- Pri upoređivanju velikog broja ulaznih sekvenci putem BLAST-a komandne linije, "megablast" je mnogo brži od višestrukog pokretanja BLAST-a. Obavlja konkatenaciju više ulaza, kako bi formirao veliku sekvencu pre pretrage, a zatim se naknadnom analizom dobijaju konačni rezultati.
Alternativne verzije[uredi | uredi izvor]
Dizajnirana verzija za poređenje velikih genoma ili DNK je BLASTZ.
CS-BLAST (ContSxt-Specific BLAST) je proširena verzija BLAST-a za pretragu proteinskih sekvenci, koja pronalazi dvostruko više daleko povezanijih sekvenci od BLAST-a za isto vrijeme i sa istom stopom greške. U CS-BLAST-a, vjerovatnoća mutacija između aminokiselina ne ovisi samo od jedne aminokiseline kao u BLAST-u, već i od konteksta lokalne sekvence. Vašingtoski univerzitet napravio je alternativnu verziju NCBI BLAST-a, zvanu WU-BLAST. Autorska prava pripadaju kompaniji Advanced Bioco, a NCBI objavio je novu seriju BLAST-ovih izvršnih programa, C++ zasnovani BLAST+,[19], i objavio je paralelnu verziju do 2.2.26. Počevši sa verzijom 2.2.27 (April 2013), dostupni su samo BLAST+ izvršni programi. Među izmjenama je i zamjena blastall komande za više različitih uputa za različite BLAST programe i promjene u rukovanju opcijama.
Alternative BLAST-a[uredi | uredi izvor]
Ekstremno brza, ali znatno manje osetljiva, alternativa BLAST-u je BLAT (eng. Blast Like Alignment Tool). Dok BLAST obavlja linearnu pretragu, BLAT se oslanja na k-mer indeksiranje baze i na tako često može brže pronaći sjeme. .[20] Još jedan program sličan BLAT-u je PatternHunter.
Napretkom tehnologija sekvenciranja, kasnih 2000-tih, nalaženje veoma sličnih nukleotida postaje važan problem. Novi programi poravnanja skovani za ovu specifičnu upotrebu koriste BWT-indeksiranje ciljane baze (obično genoma). Ulazna sekvenca može biti mapirana vrlo brzo, a izlaz je obično u vidu BAM-fajla. Primjeri programa poravnanja su BWA, SOAP i Bowtie. Za identificiranje proteina, traženje poznatih domena (npr. Pfam) povezivanjem sa Skrivenog Markovljevog modela popularna je alternativa, kao što je npr. HMMER. Alternativa BLAST-u za poređenje dvije banke sekvenci je KLAST[21] and ORIS[22]. Rezultati KLAST-a su veoma slični rezultatiima BLAST-a, ali KLAST je značajno brži i sposobniji da poredi velike skupove sekvenci sa malim utroškom memorije.
Primjena BLAST-a[uredi | uredi izvor]
BLAST se može koristiti za više različitih potreba, uključujući i identifikaciju vrsta, lociranje domena, rekonstrukciju filogeneze, DNK mapiranje i poređenje.
- Identifikacija vrsta
- upotrebomm BLAST-a, moguće je tačno identificirati ili naći odgovarajuću vrstu. To može biti korisno, naprimjer, za rad sa DNK sekvencama nepoznatih vrsta.
- Lociranje domena
- proteinske sekvence moguće je upotrebiti kao ulaz BLAST, da bi se locirali poznati domeni te sekvence.
- Uspostava filogenije
- Korištenjem rezultata BLAST-a, moguće je kreirati filogenetsko stablo putem BLAST web-stranice. Filogenija zasnovana samo na BLAST-u je manje pouzdana nego drugi, namjenski kreiranei, filogenetski metodi, tako da bi trebalo da bude korišćena samo kao prvi i orijentacijski produkt analize.
- Mapiranje DNK
- U radu sa poznatim vrstama i traganjz za sekvencama gena na nepoznatoj lokacijii, BLAST može porediti hromosomske pozicije sekvenci DNK.
- Poređenje
- U radovima sa genskim pokazateljima , BLAST može locirati česte gene u dvije povezane vrste, a može se koristiti i za mapiranje razlika među organizmima. Optičkoračunarski pristupi su predloženi za obećavajuće alternative sadašnjim električnim implementacijama. OptCAM je primjer takvih pristupa i pokazalo se da je brži od BLAST-a.[23]
Poređenje BLAST-a i Smith-Watermanovog procesa[uredi | uredi izvor]
Iako se i Smith-Watermanov metod i BLAST koriste za pronalaženje odgovaraćujih sekvenci pretragom i poređenje upitne sekvence sa onim iz baza, imaju određene razlike. BLAST zasnovan na heurističkom algoritmu, pa rezultati dobijeni njegovom primjrnom, u terminima broja pronađenih pogodaka, možda neće dati najbolje rezultate, jer neće pronaći sva podudaranja sa bazom. Bolja alternativa za pronalaženje najboljeg mogućeg rješenja bila bi korišćenje Smith-Watermanovog algoritma. Ovaj metod se razlikuje od BLAST-a po preciznosti i brzni. Smith-Watermanov metod obezbjeđuje veću preciznost, jer pronalazi podudaranja koja BLAST ne može, zato što ne preskače nijednu informaciju. Međutim, u poređenju sa BLAST-om, troši više vremena i zahtijeva veću količinuračunarskih resursa. Pronađene su tehnologije koje mogu znatno da ubrzaju Smith-Watermanov proces. Uključuju FPGA čipove i SIMD technologiju.
Za dobijanje boljih rezultata BLAST-a, mogu se promijeniti podrazumijevana podešavanja. Ne postoji siguran način za mijenjanje podešavanja, kako bi se obezbijedio najbolji rezultat za datu sekvencu.
Također pogledajte[uredi | uredi izvor]
Reference[uredi | uredi izvor]
- ^ Douglas Martin (21. 2. 2008). "Samuel Karlin, Versatile Mathematician, Dies at 83". The New York Times.
- ^ R. M. Casey (2005). "BLAST Sequences Aid in Genomics and Proteomics". Business Intelligence Network.
- ^ Altschul, Stephen; Gish, Warren; Miller, Webb; Myers, Eugene; Lipman, David (1990). "Basic local alignment search tool". Journal of Molecular Biology. 215 (3): 403–410. doi:10.1016/S0022-2836(05)80360-2. PMID 2231712. CS1 održavanje: nepreporučeni parametar (link)
- ^ Casey, R. M. (2005). "BLAST Sequences Aid in Genomics and Proteomics". Business Intelligence Network.
- ^ Lipman, DJ; Pearson, WR (1985). "Rapid and sensitive protein similarity searches". Science. 227 (4693): 1435–41. doi:10.1126/science.2983426. PMID 2983426.
- ^ BLAST+ executables
- ^ Index of BLAST databases (FTP)
- ^ Steven Henikoff; Jorja Henikoff (1992). "Amino Acid Substitution Matrices from Protein Blocks". PNAS. 89 (22): 10915–10919. Bibcode:1992PNAS...8910915H. doi:10.1073/pnas.89.22.10915. PMC 50453. PMID 1438297.
- ^ Mount, D. W. (2004). Bioinformatics: Sequence and Genome Analysis (2nd izd.). Cold Spring Harbor Press. ISBN 978-0-87969-712-9.
- ^ SEG
- ^ DUST
- ^ Gumbel extreme value distribution (EVD)
- ^ BLOSUM62
- ^ Yim, WC; Cushman, JC (2017). "Divide and Conquer (DC) BLAST: fast and easy BLAST execution within HPC environments". PeerJ. 5: e3486. doi:10.7717/peerj.3486. PMC 5483034. PMID 28652936.
- ^ "Program Selection Tables of the Blast NCBI web site".
- ^ Vouzis, P. D.; Sahinidis, N. V. (2010). "GPU-BLAST: using graphics processors to accelerate protein sequence alignment". Bioinformatics. 27 (2): 182–8. doi:10.1093/bioinformatics/btq644. PMC 3018811. PMID 21088027.
- ^ Liu W, Schmidt B, Müller-Wittig W (2011). "CUDA-BLASTP: accelerating BLASTP on CUDA-enabled graphics hardware". IEEE/ACM Trans Comput Biol Bioinform. 8 (6): 1678–84. doi:10.1109/TCBB.2011.33. PMID 21339531.
- ^ {{cite journal |vauthors=Zhao K, Chu X |title=G-BLASTN: accelerating nucleotide alignment by graphics processors |journal=Bioinformatics |volume=30 |issue=10 |pages=1384–91 |date=maj 2014 |pmid=24463183 |doi=10.1093/bioinformatics/btu047
- ^ Camacho, C.; Coulouris, G.; Avagyan, V.; Ma, N.; Papadopoulos, J.; Bealer, K.; Madden, T. L. (2009). "BLAST+: Architecture and applications". BMC Bioinformatics. 10: 421. doi:10.1186/1471-2105-10-421. PMC 2803857. PMID 20003500.
- ^ Kent, W. James (1. 4. 2002). "BLAT—The BLAST-Like Alignment Tool". Genome Research (jezik: engleski). 12 (4): 656–664. doi:10.1101/gr.229202. ISSN 1088-9051. PMC 187518. PMID 11932250.
- ^ Lavenier, D.; Lavenier, Dominique (2009). "PLAST: parallel local alignment search tool for database comparison". BMC Bioinformatics. 10: 329. doi:10.1186/1471-2105-10-329. PMC 2770072. PMID 19821978.
- ^ Lavenier, D. (2009). "Ordered index seed algorithm for intensive DNA sequence comparison" (PDF). 2008 IEEE International Symposium on Parallel and Distributed Processing (PDF). str. 1–8. doi:10.1109/IPDPS.2008.4536172. ISBN 978-1-4244-1693-6.
- ^ Maleki, Ehsan; Koohi, Somayyeh; Kavehvash, Zahra; Mashaghi, Alireza (2020). "OptCAM: An ultra‐fast all‐optical architecture for DNA variant discovery". Journal of Biophotonics. 13 (1): e201900227. doi:10.1002/jbio.201900227. PMID 31397961.
Dopunska literatura[uredi | uredi izvor]
- Mount, D. W. (2004). Bioinformatics: Sequence and Genome Analysis (2nd izd.). Cold Spring Harbor Press. ISBN 978-0-87969-712-9.CS1 održavanje: ref=harv (link)
- Baxevanis, Andy (2005). "Chapter 11: Assessing Pairwise Sequence Similarity: BLAST and FASTA". Bioinformatics: A Practical Guide to the Analysis of Genes and Proteins, Third Edition. New York: John Wiley & Sons.
- Wheeler, David; Bhagwat, Medha (2007). "Chapter 9: BLAST QuickStart". u Bergman, Nicholas H (ured.). Comparative Genomics Volumes 1 and 2. Methods in Molecular Biology. 395–396. Totowa, NJ: Humana Press. PMID 21250292.
- Mount, DW (1. 7. 2007). "Using the Basic Local Alignment Search Tool (BLAST)". Cold Spring Harbor Protocols. 2007 (14): pdb.top17. doi:10.1101/pdb.top17. PMID 21357135.
Vanjski linkovi[uredi | uredi izvor]
- http://blast.ncbi.nlm.nih.gov/}}
- BLAST+ executables — free source downloads
- Biological Sequence Analysis I: Andy Baxevanis' lecture from NHGRI's Current Topics in Genome Analysis series, covering contemporary areas in genomics and bioinformatics
- What's behind BLAST?: talk by Gene Myers (slides and video)
| Commons ima datoteke na temu: BLAST |



![{\displaystyle \mu ={}^{\left[\log \left(Km'n'\right)\right]}\!\!\diagup \!\!{}_{\lambda }\;}](https://wikimedia.org/api/rest_v1/media/math/render/svg/f345aa27fbc08d03c73a72bd3581e3bd26f6a7ca)













