Trolančana DNK
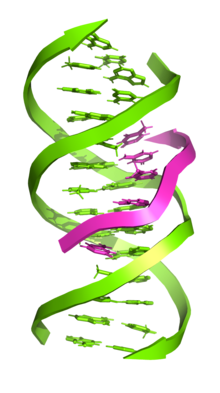
Trolančana DNK (također poznata kao H-DNK ili tripleksna-DNK) je struktura DNK u kojoj se tri oligonukleotida vijugaju jedan oko drugog i formiraju trostruku spiralu. U trolančanoj DNK, treći lanac se vezuje za DNK oblika B-DNK (preko Watson-Crickovog uparivanje baza) u dvostruku spiralu formiranjem Hoogsteenovih baznih parova ili obrnutih Hoogsteenovih vodikovih veza.
Struktura
[uredi | uredi izvor]
Primjeri trolančane DNK pronađeni su u prirodnim izvorima sa potrebnim strukturnim elementima, naprimjer u satelitskoj DNK.[1][2]
Uparivanje Hoogsteenovih baza
[uredi | uredi izvor]Timinska (T) nukleobaza može se vezati za Watson–Crickovo uparivanje baza T-A formiranjem Hoogsteenove vodikove veze. Timinski vodik se veže sa adenozinom (A) originalne dvolančane DNK, kako bi stvorio T-A*T bazni triplet.[3][4]
Međumolekulske i unutarmolekulske interakcije
[uredi | uredi izvor]
Postoje dvije klase tripleksne DNK: međumolekulske i unutarmolekulske formacije. Međumolekulski tripleks se odnosi na formiranje tripleksa između dupleksa i drugačijeg (trećeg) lanca DNK. Treći lanac može biti ili iz susjednog hromosoma ili oligonukleotida koji formira tripleks (TFO). Unutarmolekulski tripleks DNK formira se od dupleksa sa homopurinom i homopirimidinskim lancima sa simetrijom ponavljanja kao slika u ogledalu.[5] Stepen uvijanja superspirala u DNK utiče na količinu formiranja unutarmolekulskog tripleksa koja se javlja.[6] Postoje dva različita tipa unutarmolekulske tripleksne DNK: H-DNK i H*-DNK. Formiranje H-DNK se stabilizuje u kiselim uslovima i u prisustvu divalentnih kationa, kao što je Mg2+. U ovoj konformaciji, homopirimidinski lanac u dupleksu se savija unazad, kako bi se vezao za purinski lanac na paralelan način. Osnovni trolanci koji se koriste za stabilizaciju ove konformacije su T-A*T i C-G*A+. Citozin ove bazne trijade treba biti protoniran da bi se formirao ovaj unutarmolekulski trostruki heliks, zbog čega se ova konformacija stabilizuje u kiselim uslovima.[7] H*-DNK ima povoljne uslove za formiranje pri neutralnom pH i u prisustvu dvovalentnih kationa.[6] Ova unutsatarmolekulska konformacija formira se od vezivanja homopurinskog i purinskog lanca dupleksa na antiparalelni način . Stabiliziran je baznim tripletima T-A*A i C-G*G.[5][7]
Funkcija
[uredi | uredi izvor]Oligonukleotidno formiranje tripleksa (TFO)
[uredi | uredi izvor]TFO su kratki (≈15-25 nt) lanci nukleinske kiseline koji se vezuju u glavni žlijeb dvolančane DNK, kako bi formirali unutarmolekulake tripleksne strukture DNK. Postoje neki dokazi da su oni također sposobni modulirati aktivnost gena in vivo. U peptidnoj nukleinskoj kiselini (PNA), šećernofosfatna okosnica DNK zamijenjena je okosnicom nalik proteinu. PNA formiraju P-petlje dok su u interakciji sa dupleksom DNK, formirajući tripleks sa jednim lancem DNK, dok istiskuju drugi. Pretpostavlja se da se pod RecA proteinom u toku homologne rekombinacije formiraju vrlo neobične rekombinacije ili paralelne triplekse ili R-DNK.[8]
TFO se specifično vezuju za homopurin-homopirimidinske regije koje su često uobičajene u promotorskim i intronskim sekvencama gena, utičući na ćelijsku signalizaciju.[9] TFO mogu inhibirati transkripciju, vezujući se sa visokom specifičnošću za spiralu DNK, čime blokiraju vezivanje i funkciju faktora transkripcije za određene sekvence. Uvođenjem TFO-a u ćeliju (transfekcijom ili drugim sredstvima), može se kontrolisati ekspresija određenih gena.[10] Ova aplikacija ima nove implikacije u mutagenezi specifičnoj za mjesto i genskoj terapiji. U ljudskim ćelijama raka prostate, transkripcijski faktor Ets2 je pretjerano izražen i smatra se da pokreće rast i preživljavanje ćelija u takvom višku. Carbone et al. dizajnirali su TFO specifičan za sekvencu za Ets2 promotorsku sekvencu koja je smanjila ekspresiju gena i dovela do usporavanja ćelijski rast i smrti ćelije.[11] Changxian et al. su također predstavili TFO koji cilja na promotorsku sekvencu bcl-2, gena koji inhibira apoptozu.[12]
Uočena inhibicija transkripcije također može imati negativne zdravstvene efekte kao što je njena uloga u recesivnom, autosomnom genu za Friedreichovu ataksiju.[13] Kod Fredrickove ataksije, formiranje tripleksa DNK narušava ekspresiju intron 1 gena FXN. To dovodi do degeneracije nervnog sistema i kičmene moždine, ometajući kretanje udova.][14] Za borbu protiv ove tripleksne nestabilnosti, pokazalo se da proteini za popravku ekscizijom nukleotida (NER) prepoznaju i popravljaju trolančane DNK strukture, vraćajući potpunu dostupnost prethodno inhibiranog i nestabilnog gena.[15]

Peptidne nukleinske kiseline (PNA)
[uredi | uredi izvor]Peptidne nukleinske kiseline su sintetski oligonukleotidi koji se odupiru degradaciji proteazama i koriste se za induciranje popravke u regijama formiranja tripleksa specifičnih za mjesto na genomskim mjestima DNK. PNA su u stanju da se vežu sa visokim afinitetom i specifičnošću sekvence za komplementarnu sekvencu DNK putem Watson-Crickovog uparivanje baza i mogu da formiraju trostruke spirale kroz paralelne orijentacije Hoogsteenove veze sa PNA okrenutim ka 5' -kraju DNK lanca.[16] PNA-DNK tripleks je stabilan jer se PNA sastoje od neutralno nabijene pseudopeptidne kičme koja se veže za sekvencu dvolančane DNK (dsDNA)..[17] Slično kao homopirimidin u TFO, homopirimidin u PNA je u stanju da formira vezu sa komplementarnim homopurinom u ciljnoj sekvenci dsDNK. Ovi analozi DNK su u stanju da se vežu za dsDNK, iskorišćavanjem ambijentnih uslova DNK i različitih načina predviđanja prepoznavanja. Ovo se razlikuje od TFO-a koji se vezuju kroz glavni žlijeb prepoznavanjem dsDNK.[16]
Jedan od korištenih načina predviđanja prepoznavanja je dupleksna invazija.[17][18] Unutar mješovitog A–T/G–C dsDNK, sekvenca je ciljana od strane par pseudokomplementarnih (pc) PNA, koji su u stanju da se vežu za dsDNK dvostrukom invazijom kroz istovremeno stvaranje diaminopurina (D) i tiouracila (Us) koji zamjenjuju adenin i timin.[18] pc PNA par formira D–T i Us–A i G–C ili C–G Watson-Crickovu uparenu spiralu PNA-DNK sa svakim od komplementarnih lanaca DNK. Drugi oblik prepoznate dupleksne invazije na ciljanu sekvencu može se pojaviti u dsDNK koja sadrži mješovite sekvence T–C.[19] Ovaj oblik dupleksne invazije postiže se komplementarnom sekvencom homopurinskih PNA oligomera. Ovaj tripleks je formiran od hibrida PNA-DNK koji se vezuje antiparalelno s komplementarnom sekvencom DNK i rezultira pomjerenim nekomplementarnim lancem DNK.[17]
Dodatno, PNA se može modificirati tako da se formiraju tripleksne strukture "zatezanja" na ciljnom mjestu.[18] Jedan tip formirane "stezaljke" je bis-PNA struktura, u kojoj se dvije molekule PNA drže zajedno fleksibilnim linkerom kao što je 8-amino-3,6-dioksaoktanska kiselina (O).[20] Bis-PNA struktura formira PNA-DNK-PNA tripleks na ciljnom mjestu, gdje jedan lanac formira Watson-Crickove bazne parove sa DNK u antiparalelnoj orijentaciji, a drugi lanac formira Hoogsteenove bazne parove sa homopurinskim lancem DNK u DNK-PNA dupleks.[19] Repna stezaljka PNA (tcPNA) je također drugi oblik tripleksne stezaljke koja se također može formirati. TcPNA sadrže produženi rep od 5-10 bp koji formira PNA/DNK dupleks pored PNA-DNK-PNA "stezaljke". Ovo omogućava preciznije vezivanje PNA, bez potrebe za rastezanjem homopirimidina/piridina.[17] Pokazalo se da ove strukture stezanja imaju visok afinitet i specifičnost. Dodavanje ostataka lizina na jedan ili oba kraja PNA može se koristiti za povećanje ćelijske apsorpcije i vezivanja.[18]
Genetička regulacija
[uredi | uredi izvor]Trolančana DNK uključena je u regulaciju nekoliko gena. Naprimjer, gen c-myc je ekstenzivno mutiran kako bi se ispitala uloga koju tripleksna DNK, naspram linearne sekvence, ima u regulaciji gena. C-myc promotorski element, zvani element osjetljiv na nukleazu ili NSE, može formirati tandemske unutarmolekulske triplekse tipa H-DNK i ima motiv repetitivne sekvence (ACCCTCCCC)4. Mutirani NSE je ispitan na transkripcijsku aktivnost i na njegovu sposobnost formiranja intra- i intermolekulskog tripleksa. Transkripcia aktivnost mutantnih NSE može se predvidjeti sposobnošću elementa da formira H-DNK, a ne brojem ponavljanja, položajem ili brojem mutantnih baznih parova. DNK stoga može biti dinamički učesnik u transkripciji c-myc gena.[21]
Prema nekoliko objavljenih članaka, H-DNK ima sposobnost da reguliše ekspresiju gena u zavisnosti od faktora kao što su lokacija i sekvence u blizini. Iako su intergenski regioni prokariotskog genoma pokazali niske tragove H-DNK ili tripleks motiva koji se javljaju u prirodi, pokazalo se da su strukture H-DNK zastupljenije u eukariotskom genomu. Pokazalo se da je H-DNK posebno zastupljena u ćelijama sisara, uključujući ljude (1 na svakih 50.000 bp).[16] Genske sekvence uključene u regulaciju gena obično se nalaze u promotorskoj regiji eukariotskog genoma.[16]
Shodno tome, region promotora pokazao je sposobnost da formira H-DNK sa višom frekvencijom.[16] Bioinformatička analiza genoma S. cerevisiae uočila je pojavu H-DNK i drugih motiva trostruke DNK u četiri organizaciona regiona: introni, egzoni, promotorski regioni i razni regioni. Bioinformatika je prikazala ukupno 148 mogućih struktura H-DNK ili tripleta DNK. Promotorska regija je imala višu frekvenciju sa 71 trostrukom strukturom, dok su egzoni činili 57 triplatnih struktura, a introni i ostalo 2 i 18 struktura.[22]
Studije in vitro i in vivo ekspresije eukariotskog genoma rezultirale su jednim od tri rezultata: nadregulacija, podregulacija, ili bez promjene u prisustvu motiva H-DNK.[16] Kato et al. prijavili su ekspresiju pojačane regulacije lacZ, kada je H-DNK uveden u promotor B-laktamaze.[16][23] S druge strane, slična studija (Brachmachari et al.) prijavila je da nema statistički značajne inhibicije reporterskog gena lacZ kada je H-DNK umetnuta u genom COS sisarskih ćelija.[16] Iako studije sugeriraju regulaciju H-DNK, mehanizam se još uvijek istražuje. Potaman et al povezuju mehanizam regulacije gena sa interakcijama između H-DNK i TATA-kutije koja se nalazi u promotorskom regionu Na,K-ATPaze. U formacijama H-DNK, u blizini TATA-kutije, struktura H-DNK destabilizira T-A veze neophodne za transkripciju. Interferencija sa TATA-kutijom inhibira transkripcijski mehanizam i inicijaciju transkripcije koja ometa ekspresije gena.[16][24] Ostali mehanizmi povezani sa genomskom ekspresijom genetičke sekvence u prisustvu H-DNK uključuju TFO. Studije in vitro istakle su smanjenje ekspresija gena u prisustvu TFO u sisarskim ćelijama.[25] Drugi mogući mehanizam predstavili su Valentin et al., sugerirajući da 13-merni A–G motiv oligonukleotidni tripleksni kompleks (TFO kompleks) smanjuje transkripciju iRNK putem kompetitivne inhibicije.[26] Direktna inhibicija ekspresija gena iz H-DNK ključna je za mutagenezu, inhibiciju replikacije, pa čak i rekombinaciju DNK u genomu.[16]
Rekombinacija
[uredi | uredi izvor]
B) H-DNK struktura RecA, PDB: 7JY7
Pokazalo se da H-DNK motivi stimuliraju homolognu rekombinaciju različitim mehanizmima. Početne implikacije za ulogu H-DNK u rekombinaciji došle su ranih 1990-ih, kada je promatran RecA, protein rekombinacije bakterijske DNK sastavljen od trostruke spirale DNK. RecA pokazuje enzimsku aktivnost esencijalnu za rekombinaciju.[8][27] Homologna rekombinacija koja uključuje motive H-DNK također je pronađena kod eukariota. RadA, homologni protein RecA, pokazalo se da ima istu enzimsku aktivnost u rekombinaciji kao i RecA.[28] Protein ima sposobnost promoviranja i izmjene homolognih lanaca putem paralelnog trolančanog heliksa.[29][30] Jednolančana DNK (ssDNK) i komplementarna dvolančana DNK (dsDNK) će formirati strukturu D-petlje.[16][31] Ostali mogući mehanizami za RecA uključuji ssDNK iz dvije odvojene H-DNK strukture za formiranje Watson-Crickovih baznih parova. Nova struktura poznata je kao Hollidayeva spona, intermedijer u homolognoj rekombinaciji.[16] H-DNK se također nalazi u drugim oblicima rekombinacije. U ćelijama sisara, H-DNK sekvence su pokazale visoku učestalost rekombinacije. Naprimjer, studija provedena na ćelijskoj liniji mijeloma miševa pronašla je strukture H-DNK u Cγ2a i Cγ2b, koje učestvuju u razmjeni sestrinskih hromatida.[16]
Biološke implikacije
[uredi | uredi izvor]Genetička nestabilnost
[uredi | uredi izvor]Značajna istraživanja usmjerena su na biološke implikacije koje se odnose na prisustvo H-DNK u glavnim područjima prijeloma (Mbr) i dvolančanim tačkama prekida određenih gena. Nedavni radovi povezuju prisustvo neB-DNK struktura sa slučajevima genetičke nestabilnosti.[32]
Sekvence koje formiraju H-DNK koje se ponavljaju u polipurinu su pronađene u blizini P1 promotora gena c-MYC i povezane su sa glavnim žarišnim tačkama ovog regiona. Slučajevi genetičke nestabilnosti su također primijećeni kod F1 potomaka transgenih miševa nakon ugradnje sekvenci koje formiraju ljudsku H-DNK uparene sa sekvencama Z-DNK u njihove genome, gdje nije ranije prijavljena nikakva nestabilnost.[33] Osim toga, formiranje R.R.Y. Konformacije H-DNK su uočene na Mbr gena bcl-2. Pretpostavlja se da formiranje ovih struktura uzrokuje translokaciju t(14;18) uočenu kod mnogih karcinoma i većine folikulskih limfoma. Ovo zapažanje je dovelo do istraživanja koje je pokazalo da se značajno smanjenje translokacijskih događaja može primijetiti nakon blokiranja formiranja H-DNK, blagim promjenom sekvence ove regije.[33][34] Također je primijećeno da dugi delovi GAA·TTC formiraju veoma stabilne strukture H-DNK. Pokazalo se da interakcije između ove dvije strukture H-DNK, koje se nazivaju ljepljiva DNK, prekidaju transkripciju "X25" ili frataksinskog gena. Kako je smanjen nivo proteina frataksina povezan sa Friedreichovom ataksijom, pretpostavlja se da je formiranje ove nestabilnosti osnova za ovu genetičku bolest.[35][36] Trolančana DNK je primijećena u supernamotanoj satelitskoj DNK, u regijama gdje je broj kopija mikrosatelita vrlo varijabilan, zajedno sa strukturama Z-DNK invertiranih ponavljanja unutar veće jedinice za ponavljanje satelitske DNK od 2,1 kb. [37]
Osim toga, pokazalo se da H-DNK uzrokuje mutacije povezane s kritičnim ćelijskim procesima kao što su replikacija DNK i transkripcija.[38] Važnost ovih procesa jer je preživljavanje dovelo do razvoja složenih mehanizama popravka DNK koji omogućavaju ćelijama da prepoznaju i poprave oštećenje DNK. Nekanonske strukture DNK mogu se percipirati kao oštećenje putem ćelije, a nedavni radovi su pokazali povećanu prevalenciju mutacija u blizini sekvenci koje ne formiraju B-DNK.[38] Neke od ovih mutacija su uzrokovane interakcijama između H-DNK i enzima uključenih u replikaciju i transkripciju DNK, gdje H-DNK ometa ove procese i pokreće različite mehanizme popravka DNK. Ovo može uzrokovati genetičku nestabilnost i implicira H-DNK u formiranju raka.[38]
Pokazalo se da replikacija DNK utiče na funkciju različitih enzima za popravku DNK. Formiranje H-DNK uključuje formiranje jednolančane DNK (ssDNK), koja je podložnija napadima nukleaza.[38] Pokazalo se da različite nukleaze stupaju u interakciju s H-DNK na način ovisan o replikaciji ili replikaciji.[38]
Studija koja je koristila ljudske ćelije otkrila je da popravak ekscizijom nukleotida (NER) nukleaze ERCC1-XPF i ERCC1-XPG izaziva genetičku nestabilnost.[39] Ovi enzimi cijepaju H-DNK na petlji koju formiraju dva Hoogsteenova lanca vezana vodikom i 5' kraj drugog Watson-Crickovog lanca vodikovim vezama.[39] Za ovo cijepanje je bilo pokazalo se da indukuje velike delecije koje uzrokuju dvostruke prekide (DSB-e) u DNK što može dovesti do genetičke nestabilnosti.[38][39] U ćelijama s nedostatkom ERCC1-XPF i ERCC1-XPG, ove delecije su bile manje zastupljene u blizini sekvenci koje formiraju H-DNK.[39] Osim toga, pronađeno je više mutacija u ćelijama s nedostatkom ERCC1-XPF i ERCC1-XPG u odsustvu replikacija DNK, što sugerira da obrađuju H-DNK na način neovisno o replikacijama.[39]
Alternativno, pronađeno je da nukleaza za popravku replikacije DNK FEN1 potiskuje genetičku nestabilnost.[39] Slično tome, ERCC1-XPG, FEN1 cijepa H-DNK na 5' kraju lanca koji nije uključen u Hoogsteenovu vodikovu vezu.[39] HeLa ćelije sa nedostatkom FEN1 pokazale su veću prevalenciju delecija u blizini sekvenci koje formiraju H-DNK, ali je H-DNK-indukovana mutageneza bila izraženija u ćelijama s nedostatkom FEN1, u prisustvu replikacije DNK.[39] Ovo sugerira da FEN1 potiskuje mutagenezu izazvanu H-DNK, na način ovisan o replikaciji.[39]
H-DNK je implicirana u etiologiji raka kod ljudi zbog prevalencije sekvenci koje formiraju H-DNK blizu graničnih tačaka translokacije u genomima raka.[39] Aktivnost nukleaza, posredovana replikacijom s H-DNK ističe još jedan način mutageneze izazvane H-DNK i dovodi do rasta učestalosti raka.
Transkripcija
[uredi | uredi izvor]Sekvence koje formiraju H-DNK također mogu uzrokovati genetičku nestabilnost tako što ometaju i prerano zaustavljaju transkripciju.[38] Odmotavanje DNK uključeno u transkripciju čini ga podložnijim oštećenjima. U transkripcijski spojenim popravcima (TCR), lezija na lancu šablona DNK zaustavlja funkciju RNK-polimeraze i signalizira TCR faktorima da riješe oštećenje, tako što ga izrezuju.[40] H-DNK može se shvatiti kao jedna od ovih lezija.
Studija koja je posmatra-a transkripciju pomoću T7 RNK polimeraza na stabilnom analogu sekvence koja formira H-DNK otkrila je blokadu transkripcije na spoju dupleks-tripleks. Ovdje je šablonski lanac bio središnji lanac H-DNK, a poteškoće da se prekinu njegove Watson-Crickove i Hoogsteenove vodikove veze zaustavile su transkripciju.[41]
Kada je uočena transkripcija pomoću T7 na P0 promotoru gena c-MYC, pronađeni skraćeni produkti transkripcije ukazali su na to da je transkripcija zaustavljena u neposrednoj blizini sekvence koja formira H-DNK, nizvodno od promotora. Formiranje H-DNK u ovoj regiji sprečava T7 da putuje niz lanac šablona zbog sterne smetnje koju uzrokuje. Ovo zaustavlja transkripciju i signalizira da TCR faktori razriješe H-DNK, što rezultira ekscizijom DNK koja može uzrokovati genetičku nestabilnost.[40] Simetrija kao slika u ogledalu i prevalencija ostataka guanina u genu c -MYC daje mu veliku sklonost kanonskom formiranju strukture DNK.[42] Ovo, zajedno sa aktivnošću TCR faktora tokom transkripcije, čini ga veoma mutagenim, pri čemu ima ulogu u razvoju Burkittovog limfoma i leukemija.[40][42]
Aplikacije
[uredi | uredi izvor]Trolančani regioni DNK mogu se generirati povezivanjem oligonukleotida koji formiraju tripleks (TFO) i peptidnih nukleinskih kiselina (PNA). Historijski gledano, pokazalo se da vezivanje TFO inhibira transkripciju, replikaciju i vezivanje proteina za DNK.[18] Pokazalo se da TFO-ovi vezani za mutagene podstiču oštećenje DNK i induciraju mutagenezu.[16] Iako je poznato da TFO ometa transkripciju i replikaciju DNK, nedavne studije su pokazale da se TFO može koristiti za posredovanje modifikacije gena specifične za lokaciju i in vitro i in vivo.[18] Još jedna nedavna studija je također pokazala da se TFO mogu koristiti za supresiju onkogena i protoonkogena za smanjenje rast ćelija raka. Naprimjer, nedavna studija koristila je TFO za smanjenje učestalosti ćelijskee smrti u ćelijama hepatoma, smanjenjem ekspresije MET.
PNA TFO imaju sposobnost da pojačaju frekvencije rekombinacije, što dovodi do ciljanog, specifičnog editiranja gena. PNA-DNK-PNA tripleksni heliks je u stanju da se prepozna pomoću vlastitog ćelijskog mehanizma za popravku DNK, koji senzibilizira okolnu DNK za homolognu rekombinaciju. Da bi struktura PNA specifična za mjesto posredovala u rekombinaciji unutar sekvence DNK, struktura bis-PNA može biti spojena sa 40nt DNK fragmentom koji je homologan susjednoj regiji na ciljnom genu.[19] Pokazalo se da povezivanje TFO-a sa donorskim lancem DNK inducira rekombinaciju ciljanog gena i susjedne ciljne genske regije.[43] Mehanizam za ovaj oblik rekombinacije i popravkka je povezan sa popravkom ekscizijom nukleotida (NER) putem koji ima ulogu u prepoznavanju i popravljanju tripleksnih struktura.[18][19] Višestruka istraživanja sugeriraju da grupa A xeroderma pigmentosum (XPA) i protein replikacije A (RPA), koji su NER faktori, mogu se specifično vezati kao kompleks za umrežene tripleksne strukture. Poznato je da ovaj mehanizam, pored ostalih, igma ulogu u prepoznavanju i popravljanju tripleksnih struktura.
Isporuka TFO in vivo bila je glavna prepreka u korištenju TFO za modifikaciju gena.[44] Jedna studija o in vivo ciljanju hematopoetskih matičnih ćelija predložila je novu tehniku konjugacije PNA molekula sa peptidom koji prodire u ćelije (CPP) uz poli(mliječnu-ko-glikolnu kiselinu) (PLGA) nanočestica koje omogućavaju modifikacije od 6 bp u genu CCR5.[43] Editiranje gena CCR5 povezano je sa otpornošću na HIV-1.[45] CPP su proteini koji su u stanju da uspješno prenesu "teret", kao što su mali proteini ili molekule u ćelije. PGLA su biorazgradivi materijal koji inkapsulira PNA molekule kao nanočestice za specifične modifikacije genoma.[43] Studija je otkrila da su nanočestice PNA-DNK PGLA bile u stanju da efikasno uređuju hematopoetske matične ćelije sa nižom toksičnošću i bez virusa i konjugacija sa CPP je ponudila direktno ciljanje gena za mutagenezu specifičnu za mesto u matičnim ćelijama.
U novoj studiji genske terapije cistaste fibroze (CF), tri peptidne nukleinske kiseline (PNA) uz molekulu donora DNK su konstruirane tako da se isporučuju nanočesticama kako bi ispravile mutacije F508 del na transmembranskom regulatoru provodljivosti cistaste fibroze (CFTR) u ljudskim epitelnim ćelijama bronhija in vivo i in vitro.[46] Mutacija F508 del je mutacija koja se najčešće javlja zbog koje osoba ima cistastu fibrozu.[47] Mutacija F508 dovodi do gubitka funkcije CFTR, koji je hloridni kanal plazmamembrane koji je reguliran cikličkim adenozin-monofosfatom (cAMP). U ovoj studiji, uspjeli su stvoriti novi pristup liječenju CF, korištenjem nanočestica za ispravljanje mutacije F508 del CFTR, kako in vitro u ljudskim epitelnim ćelijama bronhija (HBE), tako i in vivo u modelu CF miša, što je rezultiralo pojavom CFTR-ovisnog transporta hlorida.
Historija
[uredi | uredi izvor]
Trolančane strukture DNK bile su uobičajene hipoteze 1950-ih, kada su se naučnici borili da otkriju pravi strukturni oblik DNK. Watson i Crick (koji su kasnije dobili Nobelovu nagradu za svoj model sa dvostrukom spiralom) su prvobitno smatrali modelom trostrukog heliksa, kao i Pauling i Corey, koji su objavili prijedlog za njihov model trostruke spirale 1953.,[48][49] kao i kolega naučnik Frejzer.[50] Međutim, Watson i Crick su ubrzo identificirali nekoliko problema s ovim modelima:
- Negativno nabijeni fosfati u blizini ose se odbijaju, ostavljajući pitanje kako trolančana struktura ostaje zajedno.
- U modelu trostruke spirale (posebno Paulingov i Coreyjev model), čini se da su neke od van der Waalsovih udaljenosti premale.
Frejzerov model se razlikovao od Paulingovog i Korijevog po tome što su u njegovom modelu fosfati spolja, a baze iznutra, međusobno povezane vodikovim vezama. Međutim, Watson i Crick su otkrili da je Fraserov model previše loše definiran da bi konkretno komentirali njegove neadekvatnosti.
Alternativna trolančana struktura DNK opisana je 1957.[51] Felsenfeld, Davies i Rich su predvidjeli da ako jedan lanac sadrži samo purine, a drugi lanac samo purine, lanac će doživjeti konformacijsku promjenu kako bi se formirala trolančana spirala DNK. Predviđeno je da se trolančana DNK (H-DNK) sastoji od jednog polipurinskska i dva polipirimidinska lanca.[8][51] Smatralo se da se javlja samo u jedan in vivo biološki proces: kao međuproizvod tokom delovanja E. coli, rekombinacijski enzim RecA.[51] Rani modeli iz 1960-ih predviđali su formiranje kompleksa između policetiilnih i guaninskih oligonukleotida. Modeli su predložili interakcije poznate kao Hoogstenovo uparivanje (ne-Watson-Crickovr interakcije) koje se nalaze u glavnom žlijebu.[8] Ubrzo nakon toga, predviđene su trostruke spirale sastavljene od jednog pirimidinskog i dva purinska lanca.<ref. name=":10"/> Otkriće dionica H-DNK u supersmotanim plazmidima dovelo je do vrhunca modernog interesa za potencijalnu funkciju tripleksnih struktura u živim sćelijama.[52] Dodatno, ubrzo je otkriveno da homopirimidin i neki oligonukleotidi bogati purinom mogu formirati stabilnu H-DNK strukturu sa strukturama specifičnim za sekvencu vezivanja homopurin-homopirimidina na dupleksima DNK.[53]
Reference
[uredi | uredi izvor]- ^ Fowler, R.F.; Skinner, D. M. (1986-07-05). "Eukariotska DNK divergira na dugom i složenom pirimidin:purinom traktu koji može usvojiti izmijenjene konformacije." The Journal of Biological Chemistry. 261 (19): 8994–9001. PMID 3013872
- ^ Fowler, R. F.; Skinner, D. M. (1986-07-05). "Eukaryotic DNA diverges at a long and complex pyrimidine:purine tract that can adopt altered conformations". The Journal of Biological Chemistry. 261 (19): 8994–9001. PMID 3013872.
- ^ Rhee S, Han Z, Liu K, Miles HT, Davies DR (decembar 1999). "Structure of a triple helical DNA with a triplex-duplex junction". Biochemistry. 38 (51): 16810–5. doi:10.1021/bi991811m. PMID 10606513.
- ^ Mergny JL, Sun JS, Rougée M, Montenay-Garestier T, Barcelo F, Chomilier J, Hélène C (oktobar 1991). "Sequence specificity in triple-helix formation: experimental and theoretical studies of the effect of mismatches on triplex stability". Biochemistry (jezik: engleski). 30 (40): 9791–8. doi:10.1021/bi00104a031. PMID 1911764.
- ^ a b Ussery DW, Sinden RR (juni 1993). "Environmental influences on the in vivo level of intramolecular triplex DNA in Escherichia coli". Biochemistry. 32 (24): 6206–13. doi:10.1021/bi00075a013. PMID 8512930.
- ^ a b Dayn A, Samadashwily GM, Mirkin SM (decembar 1992). "Intramolecular DNA triplexes: unusual sequence requirements and influence on DNA polymerization". Proceedings of the National Academy of Sciences of the United States of America. 89 (23): 11406–10. Bibcode:1992PNAS...8911406D. doi:10.1073/pnas.89.23.11406. PMC 50559. PMID 1454828.
- ^ a b Lyamichev VI, Mirkin SM, Frank-Kamenetskii MD (februar 1986). "Structures of homopurine-homopyrimidine tract in superhelical DNA". Journal of Biomolecular Structure & Dynamics. 3 (4): 667–9. doi:10.1080/07391102.1986.10508454. PMID 3271043.
- ^ a b c d Frank-Kamenetskii MD, Mirkin SM (1. 1. 1995). "Triplex DNA structures". Annual Review of Biochemistry. 64: 65–95. doi:10.1146/annurev.bi.64.070195.000433. PMID 7574496. S2CID 21426188.
- ^ Brázdová M, Tichý V, Helma R, Bažantová P, Polášková A, Krejčí A, et al. (2016). "p53 Specifically Binds Triplex DNA In Vitro and in Cells". PLOS ONE. 11 (12): e0167439. Bibcode:2016PLoSO..1167439B. doi:10.1371/journal.pone.0167439. PMC 5131957. PMID 27907175.
- ^ Graham MK, Brown TR, Miller PS (april 2015). "Targeting the human androgen receptor gene with platinated triplex-forming oligonucleotides". Biochemistry. 54 (13): 2270–82. doi:10.1021/bi501565n. PMID 25768916.
- ^ Carbone GM, Napoli S, Valentini A, Cavalli F, Watson DK, Catapano CV (3. 8. 2004). "Triplex DNA-mediated downregulation of Ets2 expression results in growth inhibition and apoptosis in human prostate cancer cells". Nucleic Acids Research. 32 (14): 4358–67. doi:10.1093/nar/gkh744. PMC 514370. PMID 15314206.
- ^ Shen C, Rattat D, Buck A, Mehrke G, Polat B, Ribbert H, et al. (februar 2003). "Targeting bcl-2 by triplex-forming oligonucleotide--a promising carrier for gene-radiotherapy". Cancer Biotherapy & Radiopharmaceuticals. 18 (1): 17–26. doi:10.1089/108497803321269296. PMID 12667305.
- ^ Sakamoto N, Chastain PD, Parniewski P, Ohshima K, Pandolfo M, Griffith JD, Wells RD (april 1999). "Sticky DNA: self-association properties of long GAA.TTC repeats in R.R.Y triplex structures from Friedreich's ataxia". Molecular Cell. 3 (4): 465–75. doi:10.1016/s1097-2765(00)80474-8. PMID 10230399.
- ^ Bacolla A, Wells RD (april 2009). "Non-B DNA conformations as determinants of mutagenesis and human disease". Molecular Carcinogenesis. 48 (4): 273–85. doi:10.1002/mc.20507. PMID 19306308. S2CID 5493647.
- ^ Kaushik Tiwari M, Adaku N, Peart N, Rogers FA (septembar 2016). "Triplex structures induce DNA double strand breaks via replication fork collapse in NER deficient cells". Nucleic Acids Research. 44 (16): 7742–54. doi:10.1093/nar/gkw515. PMC 5027492. PMID 27298253.
- ^ a b c d e f g h i j k l m n Jain A, Wang G, Vasquez KM (august 2008). "DNA triple helices: biological consequences and therapeutic potential". Biochimie. 90 (8): 1117–30. doi:10.1016/j.biochi.2008.02.011. PMC 2586808. PMID 18331847.
- ^ a b c d Hansen ME, Bentin T, Nielsen PE (juli 2009). "High-affinity triplex targeting of double stranded DNA using chemically modified peptide nucleic acid oligomers". Nucleic Acids Research. 37 (13): 4498–507. doi:10.1093/nar/gkp437. PMC 2715256. PMID 19474349.
- ^ a b c d e f g Ricciardi AS, McNeer NA, Anandalingam KK, Saltzman WM, Glazer PM (2014). "Targeted genome modification via triple helix formation". u Wajapeyee N (ured.). Cancer Genomics and Proteomics. Methods in Molecular Biology. 1176. New York, NY: Springer New York. str. 89–106. doi:10.1007/978-1-4939-0992-6_8. ISBN 978-1-4939-0991-9. PMC 5111905. PMID 25030921.
- ^ a b c d Rogers FA, Vasquez KM, Egholm M, Glazer PM (decembar 2002). "Site-directed recombination via bifunctional PNA-DNA conjugates". Proceedings of the National Academy of Sciences of the United States of America. 99 (26): 16695–700. Bibcode:2002PNAS...9916695R. doi:10.1073/pnas.262556899. PMC 139206. PMID 12461167.
- ^ Montazersaheb S, Hejazi MS, Nozad Charoudeh H (novembar 2018). "Potential of Peptide Nucleic Acids in Future Therapeutic Applications". Advanced Pharmaceutical Bulletin. 8 (4): 551–563. doi:10.15171/apb.2018.064. PMC 6311635. PMID 30607328.
- ^ Firulli AB, Maibenco DC, Kinniburgh AJ (april 1994). "Triplex forming ability of a c-myc promoter element predicts promoter strength". Archives of Biochemistry and Biophysics. 310 (1): 236–42. doi:10.1006/abbi.1994.1162. PMID 8161210.
- ^ Zain R, Sun JS (maj 2003). "Do natural DNA triple-helical structures occur and function in vivo?". Cellular and Molecular Life Sciences. 60 (5): 862–70. doi:10.1007/s00018-003-3046-3. PMID 12827276. S2CID 6227195.
- ^ Kato M, Shimizu N (oktobar 1992). "Effect of the potential triplex DNA region on the in vitro expression of bacterial beta-lactamase gene in superhelical recombinant plasmids". Journal of Biochemistry. 112 (4): 492–4. doi:10.1093/oxfordjournals.jbchem.a123927. PMID 1491004.
- ^ Potaman VN, Ussery DW, Sinden RR (juni 1996). "Formation of a combined H-DNA/open TATA box structure in the promoter sequence of the human Na,K-ATPase alpha2 gene". The Journal of Biological Chemistry. 271 (23): 13441–7. doi:10.1074/jbc.271.23.13441. PMID 8662935. S2CID 46550975.
- ^ Seidman MM, Glazer PM (august 2003). "The potential for gene repair via triple helix formation". The Journal of Clinical Investigation. 112 (4): 487–94. doi:10.1172/JCI19552. PMC 171401. PMID 12925687.
- ^ Rapozzi V, Cogoi S, Spessotto P, Risso A, Bonora GM, Quadrifoglio F, Xodo LE (januar 2002). "Antigene effect in K562 cells of a PEG-conjugated triplex-forming oligonucleotide targeted to the bcr/abl oncogene". Biochemistry. 41 (2): 502–10. doi:10.1021/bi011314h. PMID 11781088.
- ^ Bertucat G, Lavery R, Prévost C (decembar 1998). "A model for parallel triple helix formation by RecA: single-single association with a homologous duplex via the minor groove". Journal of Biomolecular Structure & Dynamics. 16 (3): 535–46. doi:10.1080/07391102.1998.10508268. PMID 10052612.
- ^ Chen J, Tang Q, Guo S, Lu C, Le S, Yan J (septembar 2017). "Parallel triplex structure formed between stretched single-stranded DNA and homologous duplex DNA". Nucleic Acids Research. 45 (17): 10032–10041. doi:10.1093/nar/gkx628. PMC 5622322. PMID 28973442.
- ^ Camerini-Otero RD, Hsieh P (april 1993). "Parallel DNA triplexes, homologous recombination, and other homology-dependent DNA interactions". Cell. 73 (2): 217–23. doi:10.1016/0092-8674(93)90224-e. PMID 8477443. S2CID 34585948.
- ^ Bertucat G, Lavery R, Prévost C (septembar 1999). "A molecular model for RecA-promoted strand exchange via parallel triple-stranded helices". Biophysical Journal. 77 (3): 1562–76. Bibcode:1999BpJ....77.1562B. doi:10.1016/S0006-3495(99)77004-9. PMC 1300444. PMID 10465767.
- ^ Yang H, Zhou C, Dhar A, Pavletich NP (oktobar 2020). "Mechanism of strand exchange from RecA-DNA synaptic and D-loop structures". Nature. 586 (7831): 801–806. Bibcode:2020Natur.586..801Y. doi:10.1038/s41586-020-2820-9. PMC 8366275 Provjerite vrijednost parametra
|pmc=(pomoć). PMID 33057191. S2CID 222349650. - ^ McKinney JA, Wang G, Mukherjee A, Christensen L, Subramanian SH, Zhao J, Vasquez KM (januar 2020). "Distinct DNA repair pathways cause genomic instability at alternative DNA structures". Nature Communications. 11 (1): 236. Bibcode:2020NatCo..11..236M. doi:10.1038/s41467-019-13878-9. PMC 6957503. PMID 31932649.
- ^ a b Wang G, Vasquez KM (juli 2014). "Impact of alternative DNA structures on DNA damage, DNA repair, and genetic instability". DNA Repair. 19: 143–51. doi:10.1016/j.dnarep.2014.03.017. PMC 4216180. PMID 24767258.
- ^ Raghavan SC, Chastain P, Lee JS, Hegde BG, Houston S, Langen R, et al. (juni 2005). "Evidence for a triplex DNA conformation at the bcl-2 major breakpoint region of the t(14;18) translocation". The Journal of Biological Chemistry. 280 (24): 22749–60. doi:10.1074/jbc.M502952200. PMID 15840562.
- ^ Wang G, Vasquez KM (septembar 2004). "Naturally occurring H-DNA-forming sequences are mutagenic in mammalian cells". Proceedings of the National Academy of Sciences of the United States of America. 101 (37): 13448–53. Bibcode:2004PNAS..10113448W. doi:10.1073/pnas.0405116101. PMC 518777. PMID 15342911.
- ^ Vetcher AA, Napierala M, Iyer RR, Chastain PD, Griffith JD, Wells RD (oktobar 2002). "Sticky DNA, a long GAA.GAA.TTC triplex that is formed intramolecularly, in the sequence of intron 1 of the frataxin gene". The Journal of Biological Chemistry. 277 (42): 39217–27. doi:10.1074/jbc.M205209200. PMID 12161437.
- ^ Fowler, R. F.; Skinner, D. M. (5. 7. 1986). "Eukaryotic DNA diverges at a long and complex pyrimidine:purine tract that can adopt altered conformations". The Journal of Biological Chemistry. 38 (1–3): 145–152. PMID 3013872.
- ^ a b c d e f g Wang G, Vasquez KM (januar 2017). "Effects of Replication and Transcription on DNA Structure-Related Genetic Instability". Genes. 8 (1): 17. doi:10.3390/genes8010017. PMC 5295012. PMID 28067787.
- ^ a b c d e f g h i j Zhao J, Wang G, Del Mundo IM, McKinney JA, Lu X, Bacolla A, et al. (januar 2018). "Distinct Mechanisms of Nuclease-Directed DNA-Structure-Induced Genetic Instability in Cancer Genomes". Cell Reports. 22 (5): 1200–1210. doi:10.1016/j.celrep.2018.01.014. PMC 6011834. PMID 29386108.
- ^ a b c Belotserkovskii BP, De Silva E, Tornaletti S, Wang G, Vasquez KM, Hanawalt PC (novembar 2007). "A triplex-forming sequence from the human c-MYC promoter interferes with DNA transcription". The Journal of Biological Chemistry. 282 (44): 32433–41. doi:10.1074/jbc.M704618200. PMID 17785457. S2CID 24211097.
- ^ Pandey S, Ogloblina AM, Belotserkovskii BP, Dolinnaya NG, Yakubovskaya MG, Mirkin SM, Hanawalt PC (august 2015). "Transcription blockage by stable H-DNA analogs in vitro". Nucleic Acids Research. 43 (14): 6994–7004. doi:10.1093/nar/gkv622. PMC 4538819. PMID 26101261.
- ^ a b Del Mundo IM, Zewail-Foote M, Kerwin SM, Vasquez KM (maj 2017). "Alternative DNA structure formation in the mutagenic human c-MYC promoter". Nucleic Acids Research. 45 (8): 4929–4943. doi:10.1093/nar/gkx100. PMC 5416782. PMID 28334873.
- ^ a b c McNeer NA, Schleifman EB, Cuthbert A, Brehm M, Jackson A, Cheng C, et al. (juni 2013). "Systemic delivery of triplex-forming PNA and donor DNA by nanoparticles mediates site-specific genome editing of human hematopoietic cells in vivo". Gene Therapy. 20 (6): 658–69. doi:10.1038/gt.2012.82. PMC 3713483. PMID 23076379.
- ^ Hnedzko D, Cheruiyot SK, Rozners E (septembar 2014). "Using triple-helix-forming Peptide nucleic acids for sequence-selective recognition of double-stranded RNA". Current Protocols in Nucleic Acid Chemistry. 58: 4.60.1–23. doi:10.1002/0471142700.nc0460s58. ISSN 1934-9270. PMC 4174339. PMID 25199637.
- ^ Schleifman EB, Bindra R, Leif J, del Campo J, Rogers FA, Uchil P, et al. (septembar 2011). "Targeted disruption of the CCR5 gene in human hematopoietic stem cells stimulated by peptide nucleic acids". Chemistry & Biology. 18 (9): 1189–98. doi:10.1016/j.chembiol.2011.07.010. PMC 3183429. PMID 21944757.
- ^ McNeer NA, Anandalingam K, Fields RJ, Caputo C, Kopic S, Gupta A, et al. (april 2015). "Nanoparticles that deliver triplex-forming peptide nucleic acid molecules correct F508del CFTR in airway epithelium". Nature Communications. 6 (1): 6952. Bibcode:2015NatCo...6.6952M. doi:10.1038/ncomms7952. PMC 4480796. PMID 25914116.
- ^ Lukacs GL, Verkman AS (februar 2012). "CFTR: folding, misfolding and correcting the ΔF508 conformational defect". Trends in Molecular Medicine. 18 (2): 81–91. doi:10.1016/j.molmed.2011.10.003. PMC 3643519. PMID 22138491.
- ^ Pauling L, Corey RB (februar 1953). "A Proposed Structure For The Nucleic Acids". Proceedings of the National Academy of Sciences of the United States of America. 39 (2): 84–97. Bibcode:1953PNAS...39...84P. doi:10.1073/pnas.39.2.84. PMC 1063734. PMID 16578429.
- ^ Pauling L, Corey RB (februar 1953). "Structure of the nucleic acids". Nature. 171 (4347): 346. Bibcode:1953Natur.171..346P. doi:10.1038/171346a0. PMID 13036888. S2CID 4151877.
- ^ Fraser RD (mart 2004). "The structure of deoxyribose nucleic acid". Journal of Structural Biology. 145 (3): 184–5. doi:10.1016/j.jsb.2004.01.001. PMID 14997898.
- ^ a b c Felsenfeld G, Davies DR, Rich A (april 1957). "Formation of a three-stranded polynucleotide molecule". Journal of the American Chemical Society. 79 (8): 2023–4. doi:10.1021/ja01565a074.
- ^ Hanvey JC, Shimizu M, Wells RD (septembar 1988). "Intramolecular DNA triplexes in supercoiled plasmids". Proceedings of the National Academy of Sciences of the United States of America. 85 (17): 6292–6. Bibcode:1988PNAS...85.6292H. doi:10.1073/pnas.85.17.6292. PMC 281955. PMID 3413097.
- ^ Mirkin SM, Lyamichev VI, Drushlyak KN, Dobrynin VN, Filippov SA, Frank-Kamenetskii MD (decembar 1987). "DNA H form requires a homopurine-homopyrimidine mirror repeat". Nature. 330 (6147): 495–7. Bibcode:1987Natur.330..495M. doi:10.1038/330495a0. PMID 2825028. S2CID 4360764.
Dopunska literatura
[uredi | uredi izvor]- Rich A (decembar 1993). "DNA comes in many forms". Gene. 135 (1–2): 99–109. doi:10.1016/0378-1119(93)90054-7. PMID 8276285.
- Soyfer VN, Potaman VN (1995). Triple-Helical Nucleic Acids. New York: Springer. ISBN 978-0-387-94495-1.
- Mills M, Arimondo PB, Lacroix L, Garestier T, Hélène C, Klump H, Mergny JL (septembar 1999). "Energetics of strand-displacement reactions in triple helices: a spectroscopic study". Journal of Molecular Biology. 291 (5): 1035–54. doi:10.1006/jmbi.1999.3014. PMID 10518941.
- Watson JD, Crick FH (april 1953). "Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid". Nature. 171 (4356): 737–8. Bibcode:1953Natur.171..737W. doi:10.1038/171737a0. PMID 13054692. S2CID 4253007.

