Urođeno stacionarno noćno sljepilo
| Urođeno stacionarno noćno sljepilo | |
|---|---|
| Drugi nazivi | Kongenitalno stacionarno noćno sljepilo |
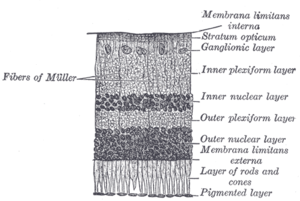 | |
| U osnovi CSNB-a je neispravnost u prijenosu od fotoreceptora u vanjskom jedarnom sloju do bipolarnih ćelija u unutarnjem jedarnom sloju. | |
| Specijalnost | Medicinska genetika, oftalmologija |
| Simptomi | Miopija, oštećen noćni vid, smanjenu oštrina vida, strabizam i nistagmus |
| Uobičajeno pojavljivanje | Prfirođena |
| Trajanje | Cjeloživotno |
| Vrste | Tip-1, tip-2 |
| Uzroci | Kvarovi u neurotransmisiji od fotoreceptora štapića i čepića do bipolarne ćelije u mrežnjači |
| Dijagnostička metoda | Elektroretinogram (ERG) |
Urođeno stacionarno noćno sljepilo (CSNB) jest rijedak neprogresivni poremećaj mrežnjača. Ljudi sa CSNB-om često imaju poteškoća s prilagodbom na situacije slabog osvjetljenja zbog oslabljene transmisije. fotoreceptora. Ovi pacijenti također mogu imati smanjenu oštrinu vida, miopiju, nistagmus i strabizam. CSNB ima dva oblika – potpuni, također poznat kao tip-1 (CSNB1) i nepotpun, također poznat kao tip-2 (CSNB2), koji se razlikuju po uključivanju različitih puteva retine. U CSNB1, nizvodni neuroni zvani bipolarne ćelije nisu u stanju da otkriju neurotransmisiju iz fotoreceptorskih ćelija. CSNB1 može biti uzrokovano mutacijama u različitim genima uključenim u detekciju neurotransmitera, uključujući NYX. U CSNB2, sami fotoreceptori imaju oštećenu funkciju neurotransmisije; ovo je prvenstveno uzrokovano mutacijama u genu CACNA1F, koji kodira naponski regulirani kalcijski kanal važan za oslobađanje neurotransmitera. CSNB je identificiran kod konja i pasa kao rezultat mutacija u TRPM1 (konji, "LP")[1], GRM6 (Konj, "CSNB2")[2] i LRIT3 (pas, CSNB).[3]
Urođeno stacionarno noćno sljepilo (CSNB) može se naslijediti po X-vezanom, autosomno dominantnom ili autosomno recesivnom obrascu, ovisno o uključenim genima.
Dva oblika CSNB-a također mogu uticati na konje, jedan je povezan s kompleksom leoparda boja dlake konja, a drugi koji se nalazi u određenim rasama konja. Oba su autosomno recesivni.[4][5]
Simptomi i znaci
[uredi | uredi izvor]X-vezane varijante kongenitalnog stacionarnog noćnog sljepila (CSNB) mogu se razlikovati od autosomnih oblika po prisutnosti miopije, koja je tipski odsutna u autosomnim oblicima. Pacijenti sa CSNB često imaju oštećen noćni vid, miopiju, smanjenu oštrinu vida, strabizam i nistagmus. Osobe sa kompletnim oblikom CSNB-a (CSNB1) imaju jako oslabljenu osjetljivost štapića (smanjena ~300x) kao i disfunkciju čunjića. Pacijenti s nekompletnom formom mogu imati ili miopiju ili hiperopiju.[6]
Uzrok
[uredi | uredi izvor]CSNB je uzrokovan kvarovima u neurotransmisiji od fotoreceptora štapića i čepiča do bipolarne ćelije u mrežnjači.[7] Kvarovi u CSNB1 specifično utiču na ON put, ometajući sposobnost bipolarnih ćelija tipa ON da detektuju neurotransmiter koji se oslobađa iz fotoreceptora.[7] Štapići, koji su odgovorni za vid pri slabom osvetljenju, ostvaruju kontakt samo sa bipolarnim ćelijama tipa ON, dok čunjići, koji su odgovorni za vid jakog svetla, ostvaruju kontakte sa bipolarnim ćelijama oba, i ON i OFF podtipa.[8] Budući da se štapići koje osvjetljava slabo svjetlo ulaze samo u ON put, osobe sa CSNB1 obično imaju problema sa noćnim vidom, dok je vid u dobro osvijetljenim uslovima pošteđen.[7] U CSNB2, oslobađanje neurotransmitera iz fotoreceptora su oštećeni, što dovodi do uključivanja i ON i OFF puteva.
Elektroretinogram (ERG) je važan alat za dijagnosticiranje CSNB. ERG a-talas, koji odražava funkciju kaskade fototransdukcija, kao odgovor na bljeskove svjetlosti, tipski je normalan kod pacijenata sa CSNB, iako je u nekim slučajevima pogođena i fototransdukcija, što dovodi do smanjenog a-talasa. ERG b-talas, koji prvenstveno odražava funkciju ON-bipolarnih ćelija, značajno je smanjen u slučajevima CSNB2, a potpuno odsutan u slučajevima CSNB1.[7][9]
Genetika
[uredi | uredi izvor]Pronađene su samo tri rodopsinske mutacije povezane s kongenitalnim stacionarnim noćnim sljepoćom (CSNB).[10] Dvije od ovih mutacija nalaze se u drugoj transmembranskoj spirali rodopsina, na Gly-90 i Thr-94. Konkretno, ove mutacije su Gly90Asp [11] i Thr94Ile, koja je posljednja prijavljena.[12] Treća mutacija je Ala292Glu, a nalazi se u sedmom transmembranskom heliksu, u blizini mjesta vezivanja mrežnjače na Lys-296.[13] Mutacije povezane sa CSNB utiču na aminokiselinske ostatke u blizini protonirane veze Schiffove baze (PSB). Povezani su s promjenama konformacijske stabilnosti i protoniranog statusa PSB dušika.[14]
Patofiziologija
[uredi | uredi izvor]CSNB1
[uredi | uredi izvor]Potpuni oblik X-vezanog kongenitalnog stacionarnog noćnog sljepila, također poznat kao niktalopija, uzrokovan je mutacijama u NYX gena (niktalopin na X-hromosomu), koji kodira mali protein porodice leucin-bogatog ponavljanja (LRR) nepoznate funkcije.[15][16] Ovaj protein sastoji se od N-terminalnog signalnog peptida i 11 LRR (LRR1-11) okruženih LRR-ovima bogatim cisteinom (LRRNT i LRRCT). Na C-terminalu proteina postoji pretpostavljeno mjesto zvano GPI-sidro. Iako funkcija NYX-a tek treba u potpunosti biti shvaćena, vjeruje se da se nalazim vanćelijski. Prirodna delecija 85 baza u NYX kod nekih miševa dovodi do fenotipa "nob" (bez b-talasa), koji je vrlo sličan onom koji se vidi kod pacijenata sa CSNB1.[17] NYX se prvenstveno eksprimira u ćelijama štapića i čunjića retine. Do sada postoji skoro 40 poznatih mutacija u NYX-u povezanih sa CSNB1 (tabela 1.), lociranih u cijelom proteinu. Kako je funkcija proteina niktalopina nepoznata, ove mutacije nisu dalje karakterizirane. Međutim, predviđa se da će mnogi od njih dovesti do skraćenih proteina koji su, vjerovatno, nefunkcionalni.
| Mutacija | Pozicija | Reference | |
|---|---|---|---|
| Nukleotid | Aminokiselina | ||
| c.?-1_?-61del | 1_20del | Signalna sekvenca | [16] |
| Prerada RNK | Intron 1 | [18] | |
| c.?-63_1443-?del | 21_481del | [16] | |
| c.48_64del | L18RfsX108 | Signalna sekvenca | [18] |
| c.85_108del | R29_A36del | N-krajnji LRR | [15] |
| c.G91C | C31S | LRRNT | [16] |
| c.C105A | C35X | LRRNT | [16] |
| c.C169A | P57T | LRRNT | [19] |
| c.C191A | A64E | LRR1 | [19] |
| c.G281C | R94P | LRR2 | [20] |
| c.301_303del | I101del | LRR2 | [16] |
| c.T302C | I101T | LRR2 | [20] |
| c.340_351del | E114_A118del | LRR3 | [16][18] |
| c.G427C | A143P | LRR4 | [16] |
| c.C452T | P151L | LRR4 | [15] |
| c.464_465insAGCGTGCCCGAGCGCCTCCTG | S149_V150dup+P151_L155dup | LRR4 | [15] |
| c.C524G | P175R | LRR5 | [16] |
| c.T551C | L184P | LRR6 | [15] |
| c.556_618delini | H186?fsX260 | LRR6 | [15] |
| c.559_560deliniAA | A187K | LRR6 | [16] |
| c.613_621dup | 205_207dup | LRR7 | [15][16] |
| c.628_629ins | R209_S210iniCLR | LRR7 | [15] |
| c.T638A | L213Q | LRR7 | [15] |
| c.A647G | N216S | LRR7 | [15][18] |
| c.T695C | L232P | LRR8 | [15] |
| c.727_738del | 243_246del | LRR8 | [16] |
| c.C792G | N264K | LRR9 | [15] |
| c.T854C | L285P | LRR10 | [15] |
| c.T893C | F298S | LRR10 | [15] |
| c.C895T | Q299X | LRR10 | [18] |
| c.T920C | L307P | LRR11 | [16] |
| c.A935G | N312S | LRR11 | [16] |
| c.T1040C | L347P | LRRCT | [16] |
| c.G1049A | W350X | LRRCT | [15] |
| c.G1109T | G370V | LRRCT | [16] |
| c.1122_1457del | S374RfsX383 | LRRCT | [16][18] |
| c.1306del | L437WfsX559 | C-kraj | [18] |
| LRR: leucin-bogato ponavljanje, LRRNT i LRRCT: N- i C-krajnji cistein-bogati LRR-i. | |||
CSNB2
[uredi | uredi izvor]
Nepotpuni oblik X-vezanog kongenitalnog stacionarnog noćnog sljepila (CSNB2) uzrokovan je mutacijama u genu CACNA1F, koji kodira naponsko-ovisni kalcijski kanal CaV1.4 eksprimiran u mrežnjačama.[21][22] Jedno od važnih svojstava ovog kanala je da se inaktivira izuzetno malom brzinom. Ovo mu omogućava da proizvodi trajni unos Ca2+ nakon depolarizacije. Kako se fotoreceptori depolariziraju u odsustvu svjetlosti, CaV1.4 kanali funkcionišu kako bi osigurali kontinuirano oslobađanje neurotransmitera nakon depolarizacije.[23] Ovo je pokazano kod CACNA1F mutantnih miševa koji su značajno smanjili fotoreceptorske kalcijeve signale.[24] Postoji 55 mutacija u CACNA1F lociranih u cijelom kanalu, tabela 2 i slika 1. Iako većina ovih mutacija rezultira skraćenim i vjerovatno nefunkcionalnim kanalima, očekuje se da one sprječavaju sposobnost svjetlosti da hiperpolarizira fotoreceptore. Od mutacija s poznatim funkcionalnim posljedicama, 4 proizvode kanale koji su ili potpuno nefunkcionalni, a dvije koje rezultiraju kanalima koji se otvaraju na daleko više hiperpolariziranih potencijala nego kod divljeg tipa. To će rezultirati fotoreceptorima koji nastavljaju oslobađati neurotransmiter čak i nakon hiperpolarizacije izazvane svjetlom.
| Mutacija | Pozicija | Efekt | Reference | |
|---|---|---|---|---|
| Nukleotid | Aminokiselina | |||
| c.C148T | R50X | N-kraj | [25] | |
| c.151_155delAGAAA | R51PfsX115 | N-kraj | [26] | |
| c.T220C | C74R | N-kraj | [26] | |
| c.C244T | R82X | N-kraj | [25][26] | |
| c.466_469delinsGTAGGGGTGCT CCACCCCGTAGGGGTGCTCCACC |
S156VdelPinsGVKHOVGVLH | D1S2-3 | [25][27][28] | |
| Prerada RNK | Intron 4 | [25] | ||
| c.T685C | S229P | D1S4-5 | [26] | |
| c.G781A | G261R | D1-pora | [26] | |
| c.G832T | E278X | D1-pora | [18][29] | |
| c.904insG | R302AfsX314 | D1-pora | [27] | |
| c.951_953delCTT | F318del | D1-pora | [25] | |
| c.G1106A | G369D | D1S6 | Aktivira ~20mV negativnije od divljeg tipa, povećava vrijeme do vršne struje i smanjuje inaktivaciju, povećava propusnost Ca2+. | [21][23][25][26][30] |
| c.1218delC | W407GfsX443 | D1-2 | [22][25][29] | |
| c.C1315T | Q439X | D1-2 | [26] | |
| c.G1556A | R519Q | D1-2 | Spanjena ekspresija | [21][31] |
| c.C1873T | R625X | D2S4 | [25][26] | |
| c.G2021A | G674D | D2S5 | [23][25][27] | |
| c.C2071T | R691X | D2-pora | [19] | |
| c.T2258G | F753C | D2S6 | [26] | |
| c.T2267C | I756T | D2S6 | Aktivira ~35mV negativnije od divljeg tipa, sporije se inaktivira | [32] |
| Prerada RNK | Intron 19 | [26] | ||
| c.T2579C | L860P | D2-3 | [26] | |
| c.C2683T | R895X | D3S1-2 | [18][19][22][25] | |
| Prerada RNK | Intron 22 | [26][27] | ||
| Prerada RNK | Intron 22 | [26] | ||
| c.C2783A | A928D | D3S2-3 | [23][25] | |
| c.C2905T | R969X | D3S4 | [21][26] | |
| c.C2914T | R972X | D3S4 | [29] | |
| Prerada RNK | Intron 24 | [25] | ||
| c.C2932T | R978X | D3S4 | [27] | |
| c.3006_3008delCAT | I1003del | D3S4-5 | [25] | |
| c.G3052A | G1018R | D3S5 | [26] | |
| c.3125delG | G1042AfsX1076 | D3-pora | [25] | |
| c.3166insC | L1056PfsX1066 | D3-pora | [21][22][25][26] | |
| c.C3178T | R1060W | D3-pora | [21][26] | |
| c.T3236C | L1079P | D3-pora | Ne otvara se bez BayK-a, aktivira ~5mV negativnije od divljeg tipa | [26][30] |
| c.3672delC | L1225SfsX1266 | D4S2 | [22][25] | |
| c.3691_3702del | G1231_T1234del | D4S2 | [21][26] | |
| c.G3794T | S1265I | D4S3 | [19] | |
| c.C3886A | R1296S | D4S4 | [19] | |
| c.C3895T | R1299X | D4S4 | [22][25][26] | |
| Prerada RNK | Intron 32 | [26] | ||
| c.C4075T | Q1359X | D4-pora | [21][26] | |
| c.T4124A | L1375H | D4-pora | Smanjena ekspresija | [21][26][31] |
| Splajsing | Intron 35 | [26] | ||
| c.G4353A | W1451X | C-kraj | Nefunkcionalan | [22][23][25][30] |
| c.T4495C | C1499R | C-kraj | [26] | |
| c.C4499G | P1500R | C-kraj | [26] | |
| c.T4523C | L1508P | C-kraj | [26] | |
| Splajsing | intron 40 | [25] | ||
| c.4581delC | F1528LfsX1535 | C-kraj | [33] | |
| c.A4804T | K1602X | C-kraj | [21][26] | |
| c.C5479T | R1827X | C-kraj | [26] | |
| c.5663delG | S1888TfsX1931 | C-kraj | [25] | |
| c.G5789A | R1930H | C-kraj | [19] | |
Reference
[uredi | uredi izvor]- ^ Bellone RR, Holl H, Setaluri V, Devi S, Maddodi N, Archer S, et al. (22. 10. 2013). "Evidence for a retroviral insertion in TRPM1 as the cause of congenital stationary night blindness and leopard complex spotting in the horse". PLOS ONE. 8 (10): e78280. Bibcode:2013PLoSO...878280B. doi:10.1371/journal.pone.0078280. PMC 3805535. PMID 24167615.
- ^ Hack YL, Crabtree EE, Avila F, Sutton RB, Grahn R, Oh A, et al. (mart 2021). "Whole-genome sequencing identifies missense mutation in GRM6 as the likely cause of congenital stationary night blindness in a Tennessee Walking Horse". Equine Veterinary Journal. 53 (2): 316–323. doi:10.1111/evj.13318. PMID 32654228. S2CID 220500585.
- ^ Das RG, Becker D, Jagannathan V, Goldstein O, Santana E, Carlin K, et al. (oktobar 2019). "Genome-wide association study and whole-genome sequencing identify a deletion in LRIT3 associated with canine congenital stationary night blindness". Scientific Reports. 9 (1): 14166. Bibcode:2019NatSR...914166D. doi:10.1038/s41598-019-50573-7. PMC 6775105. PMID 31578364.
- ^ "Appaloosa Panel 2 | Veterinary Genetics Laboratory". vgl.ucdavis.edu. Pristupljeno 11. 10. 2022.
- ^ "Congenital Stationary Night Blindness (CSNB2) in Tennessee Walking Horses | Veterinary Genetics Laboratory". vgl.ucdavis.edu (jezik: engleski). Pristupljeno 11. 10. 2022.
- ^ Boycott KM, Pearce WG, Musarella MA, Weleber RG, Maybaum TA, Birch DG, et al. (april 1998). "Evidence for genetic heterogeneity in X-linked congenital stationary night blindness". American Journal of Human Genetics. 62 (4): 865–875. doi:10.1086/301781. PMC 1377021. PMID 9529339.
- ^ a b c d Zeitz C, Robson AG, Audo I (mart 2015). "Congenital stationary night blindness: an analysis and update of genotype-phenotype correlations and pathogenic mechanisms". Progress in Retinal and Eye Research. 45: 58–110. doi:10.1016/j.preteyeres.2014.09.001. PMID 25307992. S2CID 45696921.
- ^ Dunn FA, Wong RO (novembar 2014). "Wiring patterns in the mouse retina: collecting evidence across the connectome, physiology and light microscopy". The Journal of Physiology. 592 (22): 4809–4823. doi:10.1113/jphysiol.2014.277228. PMC 4259528. PMID 25172948.
- ^ Audo I, Robson AG, Holder GE, Moore AT (2008). "The negative ERG: clinical phenotypes and disease mechanisms of inner retinal dysfunction". Survey of Ophthalmology. 53 (1): 16–40. doi:10.1016/j.survophthal.2007.10.010. PMID 18191655.
- ^ Garriga P, Manyosa J (septembar 2002). "The eye photoreceptor protein rhodopsin. Structural implications for retinal disease". FEBS Letters. 528 (1–3): 17–22. doi:10.1016/s0014-5793(02)03241-6. PMID 12297272. S2CID 41860711.
- ^ Rao VR, Cohen GB, Oprian DD (februar 1994). "Rhodopsin mutation G90D and a molecular mechanism for congenital night blindness". Nature. 367 (6464): 639–42. Bibcode:1994Natur.367..639R. doi:10.1038/367639a0. PMID 8107847. S2CID 4311079.
- ^ N. al-Jandal, G.J. Farrar, A.S. Kiang, M.M. Humphries, N. Bannon, J.B. Findlay, P. Humphries and P.F. Kenna Hum. Mutat. 13 (1999), pp. 75–81.
- ^ Dryja TP, Berson EL, Rao VR, Oprian DD (juli 1993). "Heterozygous missense mutation in the rhodopsin gene as a cause of congenital stationary night blindness". Nature Genetics. 4 (3): 280–3. doi:10.1038/ng0793-280. PMID 8358437. S2CID 7682929.
- ^ Sieving PA, Richards JE, Naarendorp F, Bingham EL, Scott K, Alpern M (januar 1995). "Dark-light: model for nightblindness from the human rhodopsin Gly-90-->Asp mutation". Proceedings of the National Academy of Sciences of the United States of America. 92 (3): 880–4. Bibcode:1995PNAS...92..880S. doi:10.1073/pnas.92.3.880. PMC 42724. PMID 7846071.
- ^ a b c d e f g h i j k l m n o Bech-Hansen NT, Naylor MJ, Maybaum TA, Sparkes RL, Koop B, Birch DG, et al. (novembar 2000). "Mutations in NYX, encoding the leucine-rich proteoglycan nyctalopin, cause X-linked complete congenital stationary night blindness". Nature Genetics. 26 (3): 319–323. doi:10.1038/81619. PMID 11062471. S2CID 10223880.
- ^ a b c d e f g h i j k l m n o p q Pusch CM, Zeitz C, Brandau O, Pesch K, Achatz H, Feil S, et al. (novembar 2000). "The complete form of X-linked congenital stationary night blindness is caused by mutations in a gene encoding a leucine-rich repeat protein". Nature Genetics. 26 (3): 324–327. doi:10.1038/81627. PMID 11062472. S2CID 42428370.
- ^ Gregg RG, Mukhopadhyay S, Candille SI, Ball SL, Pardue MT, McCall MA, Peachey NS (januar 2003). "Identification of the gene and the mutation responsible for the mouse nob phenotype". Investigative Ophthalmology & Visual Science. 44 (1): 378–384. doi:10.1167/iovs.02-0501. PMID 12506099.
- ^ a b c d e f g h i Zito I, Allen LE, Patel RJ, Meindl A, Bradshaw K, Yates JR, et al. (februar 2003). "Mutations in the CACNA1F and NYX genes in British CSNBX families". Human Mutation. 21 (2): 169. doi:10.1002/humu.9106. PMID 12552565. S2CID 13143864.
- ^ a b c d e f g Zeitz C, Minotti R, Feil S, Mátyás G, Cremers FP, Hoyng CB, Berger W (mart 2005). "Novel mutations in CACNA1F and NYX in Dutch families with X-linked congenital stationary night blindness". Molecular Vision. 11: 179–183. PMID 15761389.
- ^ a b Xiao X, Jia X, Guo X, Li S, Yang Z, Zhang Q (2006). "CSNB1 in Chinese families associated with novel mutations in NYX". Journal of Human Genetics. 51 (7): 634–640. doi:10.1007/s10038-006-0406-5. PMID 16670814.
- ^ a b c d e f g h i j Strom TM, Nyakatura G, Apfelstedt-Sylla E, Hellebrand H, Lorenz B, Weber BH, et al. (juli 1998). "An L-type calcium-channel gene mutated in incomplete X-linked congenital stationary night blindness". Nature Genetics. 19 (3): 260–263. doi:10.1038/940. PMID 9662399. S2CID 34467174.
- ^ a b c d e f g Bech-Hansen NT, Naylor MJ, Maybaum TA, Pearce WG, Koop B, Fishman GA, et al. (juli 1998). "Loss-of-function mutations in a calcium-channel alpha1-subunit gene in Xp11.23 cause incomplete X-linked congenital stationary night blindness". Nature Genetics. 19 (3): 264–267. doi:10.1038/947. PMID 9662400. S2CID 42480901.
- ^ a b c d e McRory JE, Hamid J, Doering CJ, Garcia E, Parker R, Hamming K, et al. (februar 2004). "The CACNA1F gene encodes an L-type calcium channel with unique biophysical properties and tissue distribution". The Journal of Neuroscience. 24 (7): 1707–1718. doi:10.1523/JNEUROSCI.4846-03.2004. PMC 6730460. PMID 14973233.
- ^ Mansergh F, Orton NC, Vessey JP, Lalonde MR, Stell WK, Tremblay F, et al. (oktobar 2005). "Mutation of the calcium channel gene Cacna1f disrupts calcium signaling, synaptic transmission and cellular organization in mouse retina". Human Molecular Genetics. 14 (20): 3035–3046. doi:10.1093/hmg/ddi336. PMID 16155113.
- ^ a b c d e f g h i j k l m n o p q r s t Boycott KM, Maybaum TA, Naylor MJ, Weleber RG, Robitaille J, Miyake Y, et al. (februar 2001). "A summary of 20 CACNA1F mutations identified in 36 families with incomplete X-linked congenital stationary night blindness, and characterization of splice variants". Human Genetics. 108 (2): 91–97. doi:10.1007/s004390100461. PMID 11281458. S2CID 2844173.
- ^ a b c d e f g h i j k l m n o p q r s t u v w x y z aa ab ac Wutz K, Sauer C, Zrenner E, Lorenz B, Alitalo T, Broghammer M, et al. (august 2002). "Thirty distinct CACNA1F mutations in 33 families with incomplete type of XLCSNB and Cacna1f expression profiling in mouse retina". European Journal of Human Genetics. 10 (8): 449–456. doi:10.1038/sj.ejhg.5200828. PMID 12111638.
- ^ a b c d e Nakamura M, Ito S, Terasaki H, Miyake Y (juni 2001). "Novel CACNA1F mutations in Japanese patients with incomplete congenital stationary night blindness". Investigative Ophthalmology & Visual Science. 42 (7): 1610–1616. PMID 11381068.
- ^ Nakamura M, Ito S, Piao CH, Terasaki H, Miyake Y (juli 2003). "Retinal and optic disc atrophy associated with a CACNA1F mutation in a Japanese family". Archives of Ophthalmology. 121 (7): 1028–1033. doi:10.1001/archopht.121.7.1028. PMID 12860808.
- ^ a b c Allen LE, Zito I, Bradshaw K, Patel RJ, Bird AC, Fitzke F, et al. (novembar 2003). "Genotype-phenotype correlation in British families with X linked congenital stationary night blindness". The British Journal of Ophthalmology. 87 (11): 1413–1420. doi:10.1136/bjo.87.11.1413. PMC 1771890. PMID 14609846.
- ^ a b c Hoda JC, Zaghetto F, Koschak A, Striessnig J (januar 2005). "Congenital stationary night blindness type 2 mutations S229P, G369D, L1068P, and W1440X alter channel gating or functional expression of Ca(v)1.4 L-type Ca2+ channels". The Journal of Neuroscience. 25 (1): 252–259. doi:10.1523/JNEUROSCI.3054-04.2005. PMC 6725195. PMID 15634789.
- ^ a b Hoda JC, Zaghetto F, Singh A, Koschak A, Striessnig J (mart 2006). "Effects of congenital stationary night blindness type 2 mutations R508Q and L1364H on Cav1.4 L-type Ca2+ channel function and expression". Journal of Neurochemistry. 96 (6): 1648–1658. doi:10.1111/j.1471-4159.2006.03678.x. PMID 16476079. S2CID 25987619.
- ^ Hemara-Wahanui A, Berjukow S, Hope CI, Dearden PK, Wu SB, Wilson-Wheeler J, et al. (maj 2005). "A CACNA1F mutation identified in an X-linked retinal disorder shifts the voltage dependence of Cav1.4 channel activation". Proceedings of the National Academy of Sciences of the United States of America. 102 (21): 7553–7558. Bibcode:2005PNAS..102.7553H. doi:10.1073/pnas.0501907102. PMC 1140436. PMID 15897456.
- ^ Jacobi FK, Hamel CP, Arnaud B, Blin N, Broghammer M, Jacobi PC, et al. (maj 2003). "A novel CACNA1F mutation in a french family with the incomplete type of X-linked congenital stationary night blindness". American Journal of Ophthalmology. 135 (5): 733–736. doi:10.1016/S0002-9394(02)02109-8. PMID 12719097.
